CUADERNO DE PRÁCTICAS GENETICA … · AISLAMIENTO DE DNA PLASMIDICO INTRODUCCIÓN Un elemento...
Transcript of CUADERNO DE PRÁCTICAS GENETICA … · AISLAMIENTO DE DNA PLASMIDICO INTRODUCCIÓN Un elemento...

DEPARTAMENTO DE BIOQUÍMICA Y BIOLOGÍA MOLECULAR III
FACULTAD DE MEDICINA
UNIVERSIDAD COMPLUTENSE DE MADRID
Grado en Medicina
DEPARTAMENTO DE BIOQUÍMICA Y BIOLOGÍA MOLECULAR III
FACULTAD DE MEDICINA
UNIVERSIDAD COMPLUTENSE DE MADRID
CUADERNO DE PRÁCTICAS
GENETICA MOLECULAR HUMANA
CURSO ACADÉMICO 2014/2015

INDICE
1.
Práctica #1:
Aislamiento del DNA Plasmídico
2.
Práctica #2:
La Técnica de la PCR

AISLAMIENTO DE DNA PLASMIDICO
INTRODUCCIÓN
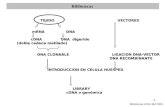
Un elemento central en la tecnología del DNA recombinante es el aislamiento y propagación dentro de un organismo (bacteria) de moléculas idénticas de DNA. Estas moléculas de DNA se copian (amplificación) utilizando la maquinaria de replicación de la célula huésped. Llamamos plásmido o vector a una molécula de DNA circular que porta la información necesaria para replicarse de forma autónoma (sin integrarse en el DNA celular) en la célula huésped. Estos plásmidos contienen genes que confieren resistencia a antibióticos, de forma que podemos seleccionar, por resistencia al antibiótico, aquellas bacterias que lo contienen frente a las que no. Otra característica importante de los plásmidos es que contienen secuencias específicas donde cortan determinadas enzimas de restricción, de forma que esto nos permite introducir en dichos sitios fragmentos de DNA de cualquier procedencia, como por ejemplo fragmentos de DNA humano, que se unirán al plásmido por medio de una ligasa. Este fragmento de DNA introducido en el vector será replicado por la célula huésped al igual que el resto del DNA de dicho vector. Este proceso de poder replicar dentro de un huésped un fragmento de DNA de otra procedencia es lo que llamamos clonaje.

El plásmido que vamos a aislar se denomina PUC18. El tamaño de este plásmido es de 2682 pares de bases y contiene el gen de la β-lactamasa, que le confiere resistencia al antibiótico ampicilina. Contiene también un sitio de inicio de replicación del DNA (sitio ori) y los genes lacI y lacZ de E. coli. Lac Z codifica para el enzima galactosidasa. Este enzima no lo contiene la cepa de E. colí que vamos a utilizar (cepa DH5α) y es capaz de transformar el sustrato artificial X-Gal en un producto coloreado de forma que aquellas bacterias que contienen el PUC18 podrían distinguirse de las que no lo contienen porque se tiñen de azul cuando ponemos X-Gal en la placa donde crecen. Pero el propósito de este enzima no es éste, ya que para ello tenemos la resistencia al antibiótico, sino que su propósito es permitirnos distinguir bacterias que contienen el plásmido PUC18 original de aquellas que contienen el plásmido con un fragmento de DNA foráneo (plásmido recombinante). Esto es así debido a que el sitio de corte de los enzimas de restricción está en la secuencia que codifica para este enzima, secuencia que queda interrumpida cuando se introduce un DNA foráneo.
MÉTODO
Preparación del gel de agarosa
Esta preparación la llevara a cabo el Profesor como una demostración a los alumnos. El gel una vez enfriado se utilizara en esta misma práctica para cargar el DNA plasmídico.
La separación de fragmentos de DNA de distinto tamaño se lleva a cabo en geles de agarosa, que se corren en cubetas horizontales. Los geles se pueden preparar con distintos porcentajes dc agarosa en función de los tamaños de los fragmentos a separar. Nosotros vamos a separar DNA plasmídico en geles de agarosa al 1 % en tampón TBE 1x (TBE: 90 mM Tris, 90 mM ácido bórico, 10 mM EDTA pH 8). La preparación del gel es como sigue:
1) Se pesa la cantidad adecuada de agarosa en polvo (0,4 g) y se añade al volumenadecuado de tampón (40 ml) TBE 1x.
2) Se calienta hasta ebullición el tiempo necesario para que se forme una solucióntransparente, y se deja enfriar durante unos minutos.
3) Se añade 4 μl de GelRed 10000X y se vierte la solución de agarosa, aun caliente yliquida, en un recipiente adecuado que contenga el denominado peine
4) Se deja transcurrir un tiempo hasta que la solución se hace un gel por enfriamiento,Se retirará el peine del gel creando así unos pocillos donde se cargarán las muestras en una solución de carga más densa que el TBE y que además contiene un colorante, azul de bromofenol, que nos marcará el frente de la carrera.

Obtención de DNA plasmídico.
1) Coger 1 mL de suspensión de bacterias E. coli que contienen el plásmido y ponerloen un tubo eppendorf.
2) Centrifugar durante l minuto en la microcentrífuga. Desechar el sobrenadante hastasequedad. Las últimas gotas sobre papel de filtro
3) Resuspender el precipitado en 300 µl de tampón TELT (50mM Tris-ClH pH 8, 62,5mM EDTA, 2,5 M LiCI, 4% Tritón X-100) para romper la pared bacteriana. Agitar suavemente
4) Añadir 600 µl de cloroformo: isopropanol (24:1). Agitar 15 segundos y centrifugar 4minutos en la microcentrífuga. De esta manera se eliminan la mayor parte de las proteínas y DNA genómico de las bacterias.
5) Recoger la fase acuosa (superior, unos 200 µl), que es la que contiene los ácidosnucleicos, y transferirla a un tubo eppendorf limpio.
6) Añadir 1 mL de etanol absoluto frío y mezclar agitando. Los ácidos nucleicos soninsolubles en etanol y esto permite su precipitación.
7) Centrifugar durante 10 minutos en la microcentrífuga, y desechar el sobrenadante.
8) Repetir los pasos 6 y 7 con 1 mL de etanol al 70% a temperatura ambiente
9) Disolver el precipitado en 10 µl de agua destilada.

Separación electroforética
1) Tomar los 10 µl de la solución de DNA plasmídico, añadir 5 µl de tampón de carga ycalentar a 70ºC durante 3 minutos para evitar la formación de agregados que no sean capaces de entrar en el gel.
2) Cargar esta muestra en el gel de agarosa y hacerla migrar a 100 voltios durante 30minutos. (Durante este tiempo se llevara a cabo el cálculo de la concentración y pureza de una muestra de DNA según se indica en este guión).
Visualización de los ácidos nucleicos.
Los fragmentos de DNA en el gel se pueden visualizar en presencia de GelRed, ya que el GelRed se intercala entre el DNA y emite una intensa fluorescencia rosa cuando se hace incidir luz ultravioleta.
La movilidad de un fragmento de DNA en un gel de agarosa es inversamente proporcional al logaritmo de su tamaño (número de pares de bases), de forma que los más pequeños migran más rápido que los más grandes.
En nuestro caso, dado que es un DNA circular obtendremos una imagen en la que se observarán más de una banda. Ello se debe a que el DNA circular puede estar relajado o superenrollado. El superenrollado migra con mayor rapidez que el relajado y el relajado a su vez dado que es un DNA circular migra un poco más lento que un DNA de igual tamaño pero lineal. La razón de todo ello es fácil de comprender si se tiene en cuenta que, por ejemplo, la molécula de DNA superenrollado es más compacta que la misma molécula relajada y por tanto puede penetrar más fácilmente a través de la trama de la agarosa. Aparte de las formas mencionadas también se observan con cierta frecuencia formas enlazadas o diméricas del plásmido. Estas serán las bandas correspondientes al plásmido y además aparecerá una banda que migra con mayor rapidez que está formada por fragmentos de degradación del RNA.
Calculo de la concentración y pureza de una muestra de DNA.
Se puede cuantificar una muestra de DNA midiendo su absorción de luz ultravioleta a 260 nm en un espectrofotómetro. Usamos cubetas eppendorf: añadimos 10 µl del DNA y 490 µl de agua estéril (volumen final 500 µl) y leemos a 260 nm. Para comprobar la posible contaminación con proteínas leemos también la absorbancia a 280 nm.

Calculo concentración: A260 x dilución muestra (50) x 50 μg/ml
Calculo pureza: A260/ A280 = 1,8 puro < 1,8 contaminación con proteínas > 1,8 contaminación con RNA
Preguntas.
1) Indicar el signo de la carga de los ácidos nucleicos y hacia que polo migrarán en uncampo eléctrico.
2) ¿Por qué un ácido nucleico de un tamaño único puede separarse en varias bandas enun gel de agarosa? Haga un esquema de las bandas que observa en el gel e interprete los resultados.
3) Indicar dos características útiles del DNA plasmídico.

UTILIZACIÓN DE LA TÉCNICA DE LA PCR (Polymerase chain reaction) PARA AMPLIFICAR UN DETERMINADO FRAGMENTO DE DNA EN EL QUE SE SOSPECHA LA EXISTENCIA DE UN POLIMORFISMO RELACIONADO CON LA DESAPARICIÓN DE UNA DIANA DE RESTRICCIÓN PARA EL ENZIMA BamHI.
Se llevaran a cabo los siguientes tres pasos: -1) Amplificar por PCR un fragmento de ~1000 pb de un DNA determinado, que se sospecha contiene el polimorfismo que sustituye una adenina por una timina en la
posición 3422 (3422 A T) lo que implica la desaparición de una diana de restricción para el enzima BamHI. Esto se traduce en una proteína anómala que tiene, en la sexta posición, una Val en vez de una Glu. A la proteína portadora de tal anomalía la denominaremos Proteína Mutada. Esta mutación puede ser la responsable de una determinada enfermedad congénita. Un caso parecido es el de la anemia falciforme, donde muta un nucleótido que forma parte de un sitio de restricción sensible a otro enzima.
-2) Incubar el producto de la PCR con el enzima de restricción BamHI
-3) Visualizar el producto de dicha incubación mediante una separación electroforética en gel de agarosa. Se observara solo una banda (~1000 pb) cuando el fragmento amplificado no sea sensible a la enzima de restricción, mientras que se observarán dos bandas de menor tamaño (de ~425 pb y de ~475 pb) si el fragmento amplificado fuera sensible al enzima de restricción BamHI. La existencia de una única banda representará la presencia del polimorfismo.

PASO 1: AMPLIFICACIÓN POR PCR Se basa en la replicación del DNA en un tubo de ensayo utilizando un termociclador con tiempo y temperatura programables. Consta de las siguientes fases:
a) Fase de desnaturalización: Para poder iniciar la replicación se requiere que las dos hebras del DNA molde se encuentren separadas, lo que puede conseguirse aumentando la temperatura del medio entre 90-95 ºC durante 3-5 min.
b) Fase de hibridación o annealing: Requiere dos cebadores o primers. Estos cebadores deben ser diseñados como secuencias complementarias de cada uno de los dos extremos 3’ de la región del DNA que se va a amplificar. Normalmente, se conocen como primer forward y primer reverse. Estos cebadores permiten a la enzima DNA polimerasa el inicio de la síntesis del DNA enlazando los desoxirribonucleotidos (dATP, dTTP, dCTP y dGTP) o dNTPs. Para la unión de los cebadores a las secuencias complementarias del DNA se requiere una bajada de la temperatura hasta aproximadamente a 50-60 ºC. Para lograrlo, la DNA polimerasa a usar debe ser termoestable. Esta característica la tiene la Taq polimerasa. La temperatura se selecciona para que el procedimiento tenga el máximo de especificidad y sensibilidad. El tiempo de hibridación de los cebadores suele estar entre 20-40 seg.
c) Fase de extensión: Se incrementará la temperatura del medio hasta alcanzar la temperatura óptima de funcionamiento de la Taq polimerasa (alrededor de los 72ºC). Los tiempos de elongación pueden estar entre 20 seg para fragmentos de 500 pb, hasta el doble para fragmentos de >1,2 Kb.

d) Fase de desnaturalización: Las hélices de DNA formadas se separaran por aumento de la temperatura a (90-95ºC) usando tiempos más cortos que la primera desnaturalización.
Las fases b) c) y d) se repiten n veces hasta alcanzar la amplificación deseada. Hay que tener en cuenta la vida media de la Taq polimerasa a altas temperaturas. También deben añadirse en cantidad suficiente los cebadores y los dNTPs.
e) Fase de extensión final: A veces, tras el último ciclo se añade un periodo de incubación a 72ºC de 5-12 min para completar la elongación de los productos que hayan quedado parcialmente sintetizados.
MATERIAL
DNA de partida (MUESTRAS 1-9): Partimos de 2 tipos de fragmentos de DNA distintos (normal y mutado). Ambos tipos contienen la secuencia de ~1000 pb que queremos amplificar. Se encuentran en disolución a una concentración de 2 ng/µL. La cantidad final añadida al tubo de reacción serán 50 ng.

Master Mix: Comprende todos los otros elementos necesarios para llevar a cabo la reacción de PCR
PROCEDIMIENTO
Cada pareja preparará 1 sólo tubo de PCR con el fin de identificar si la muestra amplificada contiene el DNA normal o el mutado.
1. Rotular un tubo de PCR con el nº de muestra elegido (del 1 al 9) 2. Añadir 25 µl del DNA problema 3. Añadir 25 µl de Master Mix al tubo de PCR. 4. Introducir en el termociclador, presionar EJECUTAR, seleccionar el programa
“practicas” en el display , presionar EJECUTAR y luego INICIAR. El programa “practicas” desarrolla el siguiente protocolo de amplificación:
Ciclos Tª Tiempo
1 94 ºC 5 min Desnaturalización inicial
33 94 ºC 30 seg Desnaturalización 62 ºC 30 seg Hibridación 72 ºC 60 seg Extensión
1 72 ºC 10 min Extensión final
Hold 4 ºC Hold Conservación de la muestra
5. Cuando finalice el programa “practicas" (± 2h) se retiran los tubos del
termociclador y se conservan a 4ºC (en la nevera) para el día siguiente.
Concentración final
Buffer 10X 1X (Contiene Mg2+ 2 mM)
dNTPs 200 µM/cada
Primers (F + R) 1 µM/cada
Taq Polimerasa 1 U
Agua MilliQ estéril --

PASO 2: CORTE CON EL ENZIMA DE RESTRICCIÓN BamHI
MATERIAL
Agua Milli Q autoclavada, para diluir el producto de la PCR.
Buffer 10X Green: es el medio en el que trabaja el enzima de restricción y, además, contiene tampón de carga. Esto permitirá cargar más adelante en un gel de agarosa, directamente, el producto final de la digestión.
Productos de amplificación del día anterior (del 1 al 9) conservados a 4ºC. Cada pareja trabajará con la muestra del mismo número que preparó para la PCR en el paso anterior.
PROCEDIMIENTO
1. Añadir 150 µl de agua Milli Q autoclavada directamente en el eppendorf de 0.2 ml, para diluir el producto de la PCR.
2. En otro tubo eppendorf de 1,5 ml preparar la digestión con el enzima de restricción:
a. Añadir:
3. Incubar 5 minutos a 37 ºC 4. Incubar 5 minutos a 80 ºC, para desactivar el enzima
Volumen
Agua MilliQ estéril 21 µl
Buffer 10X Green 3 µl
BamHI 2 µl
DNA de la PCR 4 µl

PASO 3: VISUALIZAR LOS RESULTADOS MATERIAL
Solución TBE 1X (90 mM Tris/90 mM ácido bórico/10 mM EDTA) pH 8.0
Patrón de longitud de fragmentos de DNA: Patrón comercial. Cargar 1-2 µl.
Gel de agarosa al 1% (0.4 gr de agarosa en 40 ml de TBE + 4 µl GelRed 10000X). El Gel Red es un intercalante del DNA que nos va a permitir visualizar la muestra de DNA en el gel cuando este se estimula con luz UV.
PROCEDIMIENTO
1. Cargar el producto de la reacción de digestión (10-12 µl) en un gel de agarosa al 1%.
2. Paralelamente, cargar un patrón de longitud de fragmentos de DNA ya preparado en otro pocillo (1-2 µl).
3. Correr el gel 40 minutos a 110 V. 4. Visualizar las bandas en el transiluminador
Se observara una banda de ~1000 pb cuando el fragmento amplificado no sea sensible al enzima de restricción y dos bandas de ~425 pb y de ~475 pb si el fragmento amplificado fuera sensible al enzima de restricción BamHI. La existencia de una única banda representara la presencia del polimorfismo que vamos buscando. Con el patrón podemos calcular el tamaño de la banda en pares de bases (pb).

CUESTIONES A RESOLVER 1.- Consultando la tabla de enzimas de restricción que se aporta indique el palíndromo específico para este enzima de restricción BamHI. 2.- Diga para qué se desnaturaliza la muestra inicial de DNA y como se hace. 3.-Diga de qué factores dependería el número de ciclos de la PCR. 4-Dibuje un esquema del gel resultante, que contenga un estándar de fragmentos de DNA, el fragmento no digerido y los fragmentos digeridos por BamHI.