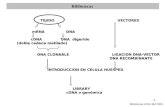
04organización del DNA
-
Upload
mischell-lazaro-ordonio -
Category
Documents
-
view
188 -
download
2
Transcript of 04organización del DNA

NIVELES DE ORGANIZACIÓN DEL DNA
MARIA CRUZ BRICEÑODTPO ACADEMICO MORFOLOGIA HUMANA FFCCMM.
UNIVERSIDAD NACIONAL DE TRUJILLO

DNA HUMANO
•DNA NUCLEAR: 99.9995 %
•DNA MITOCONDRIAL: 0.0005 %

DNA NUCLEARPares de base:3,2x109 pb (≈1.5 cm)
DNA : 3 % Codifica 97% No codifica
Cromosom as: 22 - X - Y ( 50 – 263Mb)
juegos de cromosomas por célula (óvulo/espermatozoide/ eritrocitos)
Se visualizan por: Bandeo / hibridación con sondas fluorescentes

LOS CROMOSOMAS
Son albergados en núcleos de 6 µm
Son homólogos / no homólogos (XY)
Conforman el cariotipo
Revelan perdidas o desplazamientos
Contienen genes: 25000

1 nt = mm
Genoma = 3200 Km
DNA NUCLEARCaracterística Genoma Humano
Longitud 3,2 x 109
Número de genes Aprox 25000
Gen mas grande 2,4 x 106
Tamaño medio de un gen 27000 pn
Nº mínimo de exones por gen 1
Nº máximo de exones por gen 176
Promedio de exones por gen 10,4
Tamaño de Exón mas grande 17,106 pn
Tamaño promedio de los exones 145 pb
Nº de pseudogenes Mas de 20000
% de DNA en exones 1,5 %
% de DNA altamente conservado 3,5 %
% de DNA altamente repetida 50 % aprox.

CARACTERISTICAS
Secuencias codificadoras en porcentaje pequeñoSecuencias No codificadoras; cortas , móviles , inestables en la evolución

CARACTERISTICASTamaño de los genes : 27000 pb en promedio Contienen:
Exones IntronesSecuencias reguladoras
Tienen miles de pnActiva/desactiva genes en: - Momento preciso - Nivel apropiado - Célula específica

CARACTERISTICASPresenta secuencias Conservadas
1/3 Secuencias Codifican proteínas2/3 Secuencias conservadas no codificantes
Lugares de unión a proteínas reguladorasSecuencias que producen ARNDe función desconocida

NIVELES DE ORGANIZACIÓN DEL DNA: CROMOSOMAS
INTERFASICO Se duplica Se empaqueta 500 veces
MITOTICO Se separa y reparte a
células hijas Se empaqueta 10000
veces

ELEMENTOS DEL CROMOSOMA

EMPAQUETAMIENTO DEL CROMOSOMA (DNA)

MODELO DE EMPAQUETAMIENTO
Proteínas
Histonas
No histonas
15 cm.
1,5 cm
0,3 cm..
50 μm condensado
1/3000
CROMOSOMA 1
Condensado
1/10000

NUCLEOSOMA
CROMATOSOMA DNA ESPACIADOR
80 pb
NUCLEO DEL NUCLEOSOMADNA 147 pb
1,7 vueltasHistonas: H2A , H2B, H3, H4
H1
CROMATINA 11 nm
147•DNA interfásico 30nm
Nucleosomas 11 nm

NUCLEOSOMA
HISTONAS: Proteínas pequeñas / Sec. Conservadas
Estructura: dominio de pliegue – colas terminales
PRINCIPALES PROTEINAS DE LAS HISTONAS
HISTONA PM N° DE AMINOÁCIDOS
% DE LISINA + ARGININA
H1 22500 244 30.8
H2A 13960 129 20.2
H2B 13774 125 22.4
H3 15273 135 22.9
H4 11236 102 24.5

EMSAMBLAJE DE LAS HISTONAS
NÚCLEO DEL NUCLEOSOMA
- 142 enlaces de hidrogeno entre esqueleto de aa esqueleto fosfodiester ADN
- Interacciones hidrofóbicas
- Uniones salinas

POSICIÓN DE LOS NUCLEOSOMAS
ESTÁ DETERMINADO POR:
•Secuencia del DNA
•Presencia y naturaleza de proteínas unidas al DNA

DINAMICA DE LOS NUCLEOSOMAS
Determinado por:
a. Complejos remodeladores de la cromatina dependientes de ATP
b. Proteínas con carga negativa y chaperoninas
Consecuencia:
Permite el acceso de otras proteínas al DNA nucleosómico en especial los implicados en: Expresión de los genes Replicación Reparación del DNA

a. Complejos remodeladores de la cromatina dependientes de ATP

b.Proteínas con cargas negativas – chaperoninas
Catalizan el recambio de H2A-H2B

a. Complejos remodeladores de la cromatinaMecanismo cíclico de rotura y la reorganización de los nucleosomas
Varios ciclos permiten el desplazamiento del nuclosoma

FIBRA CROMATINICA DE 30 nmLos nucleosomas se empaquetan uno sobre otro y adoptan posiciones en las que el DNA se encuentra mas condensado : mosaico fluido
Modelos:
a. Zig-Zag depende de DNA espaciador y proteínas.

b. Solenoide
Las interacciones nucleosoma –nucleosoma está dada por:Colas de histonas del nucleosoma
Histona H1 Son histonas de enlace Mas grandes y menos conservadas Provocan cambio de sentido del DNA

La fibra de 30 nm es menos uniforme ------ es dinámica
Regiones que se despliegan y se vuelven a plegar
Son regiones en transcripción en forma extendida

REGULACION DE LA ESTRUCTURA DE LA CROMATINA
• Diversas estructuras de cromatina en diferentes regiones del genoma• Herencia de las estructura cromatínicas:

Modelos de cromatina
HETEROCROMATINA
• Mas condensada• Concentrada en
telómeros, centrómeros• Mas del 10 % del genoma
de una célula• Pobre en genes • Resistente a la expresión
génica• Tipos; Constitutiva,
facultativa
EUCROMATINA• Menos condensada
– Mayormente en 30 nm lazos de 50 – 100 kb
– 10 % se presentan en estado menos condensado 10nm

b. Modificaciones covalentes de las histonas
Lugares de modificación:
• En las colas N- terminales de las histonas
Cambios:
Acetilación
Metilación
Fosforilación
Ubiquitinacón

Modificaciones en cadenas laterales en el núcleo nucleosómico
Enzima: transferasas

CONSECUENCIASAfectan la estabilidad de la fibra de 30 nm y estructuras superioresAtraer proteínas específicas que empaquetan / dan acceso al DNA
Facilita el acceso de DNA, facilita el ensamblaje de •ARN polimerasas•FT
Facilita el acceso de proteínas para empaquetar la cromatina

CROMATINA Y VARIANTES DE HISTONAS
•Histonas variantes• Se sintetizan en interfase•Se insertan en cromatina ya formada -- requieren de complejo remodelador- chaperoninas
HISTONA VARIANTE FUNCION
H3 H3.3 Activación transcripcional
CENP-A Centromérica, ensamblaje del cinetocoro
H2A H2Av Reparación y Recombinación del DNA
H2Az Expresión génica, segregación cromosomica
macroH2A Represión transcripcionalInactivación del cromosomas X

Modificaciones de variantes de Histonas variantes de Histonas produce código de histonas
Señalizan:Cromatina recién sintetizadaCromatina dañada y que necesita repararseCuando y como debe darse la expresión génica
Complejo de lectura del código
La modificaciones de H3 tiene significado específico

Complejo Lector-EscritorComplejo Lector-EscritorCambios expansivos en la cromatina
Barrera de expansión del complejo Barrera de expansión del complejo lector-escritorlector-escritor
ATPATP
ADPADP
HP1
9K-M
Onda expansiva de condensación de la cromatinaOnda expansiva de condensación de la cromatina

HETEROCROMATINA CENTROMERICA
ADN satélite α (Sec. Repetidas de 171 pb)
PROTEINAS
Histonas: metiladas poco acetiladas : Variante H3 o CEN-P A
Proteínas específicas para el centrómero --- CINETOCORO

CROMATINA CENTROMERICA – VARIANTES DE HISTONAS
CENP-A : Variante de H3Forma el nucleosoma específico del centrómero en levaduras Interacciona con proteínas específicas que lo unen al microtúbulo del cinetocoro
Centrómero en eucariotas complejos: humanoDNA satélite ó si DNA satHeterocromatina
Forman espontáneamente
El centrómero está definido porensamblaje de proteínas

ORGANIZACIÓN DE LA CORMATINA DEL CENTROMERO HUMANO
Contiene H3 NormalVariante de H3
CENP-A
CENP-A
CENP-A
H3
H3
H3


MODELO DE LA HERENCIA DE CROMATINA CENTROMERICA

Secuencias tiene la capacidad de recordar el estado de un gen, y se transmite asiHetocromatina mantiene la proteina:polycombque silencia a los genes
El empaquetamiento de DNA se da en una amplia variedad de diferentes estructura de cromatina

CROMATINA 300 nm
MAR o SAR
DEPENDE DE:
•SECUENCIAS DE UNIÓN AL SCAFFOLD Ó SECUENCIAS DE UNIÓN A LA MATRIZ
•PROTEINAS NO HISTONAS
- Condensinas (familia SMC )
- Topoisomerasa II
50000 – 200000 pn

CROMATINA INTERFASICA
Dominios cromosómicos
• Ocupan un territorio
La flourescencia revela
• Regiones ricas en genes• Regiones sin genes• Regiones intermedias

Distribución heterogénea de la cromatina:La posición de un gen en el interior del núcleo cambia cuando se expresa intensamente
Los genes se desplazan a regiones ricas en proteína necesarias para su expresión
Los genes se desplazan a regiones de silencio génico

CROMATINA 700 nm y 1400 nm
Cada molécula de DNA está organizada en bucles de cromatina (300 nmn) que procede de un esqueleto central

PROTEINAS DE LA CONDENSACION DEL CROMOSOMA MITOTICO
Condensinas :
• Utilizan ATP
•Complejos proteicos de dímeros de SMC
•SMC forman bisagras que se une al DNA
•Un dímero por cada 10000 nt de DNA
Proteínas de unión al SAR o MAR: Regiones asociadas a la matriz

Funciones del empaquetamiento de la cromatina
•Condensa el ADN individualizando las cromátidas
•Protege las frágiles moléculas de DNA cuando se separan

•SEGÚN EL GRADO DE REPETICION
SECUENCIA DE NUCLEOTIDOS DEL ADN

SECUENCIAS DE DNA
• SEGÚN EL GRADO DE REPETICIÓN
I DE ALTA REPETICIÓN:
II. DE MODERADA REPETICIÓN
III. DE SECUENCIA ÚNICA

1. ADN SATÉLITE: 106 repeticiones en tanden
ADNsat. Alfoide : >centrómeros, < disperso en el genoma
Cientos de Kb repetidas de: 4-171 pb
Motivo de 17 bases: CTTCGTTGGAAACGGGA, es reconocido por proteínas CENP-B
ADN : 68 pb: Cromosomas: 13-15, 21,22,9,1, Y
ADN Sat -1: 42 pb pericentromérico
ADN Sat-2: 5 pb
I. SECUENCIAS DE DNA DE ALTA REPETICIÓN

DNA sat

1. Retroposones:10 – 102 LINES:(L1) 6.4 Kb , bandas G
SINES(Alu)150 Mb
Pseudogenes: en familia de Globinas
2. Transposones: 10 – 102 THE humano
3. Familias génicas: 102 copias F. clásicas: ARNr, ARNt, Histonas
Superfamilias: HLA, Receptores de
células T, y hemoglobinas
4. ADN telomérico: 104
5.ADN minisatélite (VNRT): 102 - 103
6. ADN microsatelite: 104
II.SECUENCIAS DE DNA DE MODERADA REPETICION

3. ADN DE SECUENCIA UNICA
Codifican polipéptidos para
Enzimas
Hormonas
Receptores
Proteínas estructurales
Proteínas reguladoras
DNA intergenico, no repetido, función desconocida

El El acido desoxirribonucleicoacido desoxirribonucleico es la molécula que almacena la es la molécula que almacena la información de las células.información de las células.
La información contenida en La información contenida en ADNADN se expresa en se expresa en proteínasproteínas. .
Cuando se produce Cuando se produce alteraciones en el ADNalteraciones en el ADN, aparecen , aparecen proteínas proteínas alteradasalteradas, con una , con una función deficientefunción deficiente o nula, lo cual produce o nula, lo cual produce
enfermedades molecularesenfermedades moleculares