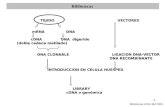
Tecnología del DNA recombinante · DNA recombinante: asociación nueva entre ... copias por...
Transcript of Tecnología del DNA recombinante · DNA recombinante: asociación nueva entre ... copias por...
Propiedad básica del DNA :
El acoplamiento de cadenas por complementariedad.La extraordinaria especificidad del reconocimientoentre secuencias de bases complementariasconstituye un poderoso instrumento para laidentificación, aislamiento, clonación, de fragmentosde DNA complementarios a uno dado
DNA recombinante: asociación nueva entrefragmentos de DNA que no se encuentranjuntos normalmente.
DNA recombinante
Secuencia para la creación de unamolécula de DNA recombinante:
1. Obtención de los fragmentos deDNA que se quieren manipular (DNAforáneo, DNA diana, DNA clonado, elinserto de DNA) utilizando enzimasde restricción
2. Unión de los segmentos de DNA
con otras moléculas denominadasvectores de clonación
3. Introducción del DNArecombinante en célulashospedadoras, donde se replicará yproducirá numerosas copias idénticaso clones
RR R
Digestió amb l'enzim de restricció
Unió
Plasmidi recombinant
Transformació de bacteris
Cromosoma bacterià
Cultiu bacterià amb moltes còpies del DNA clonat
Plasmidi
R
DNA recombinante
Molécula A Molécula B
Digestión de ambas moléculas con la misma enzima de restricción, BamHI
Mezclar
Tratar con ADN-ligasa
ADN recombinante
Extremos cohesivos
Tecnología del DNA recombinante
HERRRAMIENTAS: ENZIMAS DE
RESTRICCION
• Descubiertas por W. Arber, H. Smith y Nathans a
fines de los 60
• Reconocen secuencias específicas en el DNA de
doble cadena
• Cortan ambas cadenas en lugares concretos
• Reconocen secuencias específicas de 4-8 pares
de bases
Tipos de enzimas de restricción
• Tipo I: Realizan corte y metilación, requieren de ATP, cortan sitios aleatoriamente,distintos del sitio de reconocimiento
• Tipo II: Realizan corte, no requieren ATP, cortan dentro del sitio de reconocimiento
• Tipo III: Corte y metilación, utilizan ATP,cortan sitios aleatorios cerca de los sitios de reconocimiento
Tecnología del DNA recombinante
Clonación: vectores y hospedadores
Para conservar y propagar los fragmentos de DNAen elinterior de las células hospedadoras, es necesario emplearunos vehículos que los transporten: vectores declonación.
Características:
-Capacidad para replicarse autónomamente en el interiorde las células hospedadoras
-Capacidad para aceptar fragmentos de ácido nucleicoheterólogo, el cual se propaga y mantiene junto al vector
-Poseen un marcador seleccionable que facilita laidentificación de las células dónde ha insertado
-Su recuperación de las células hospedadoras no ha deimplicar ninguna dificultad
Tecnología del DNA recombinante
Clonación: vectores y hospedadores
VECTORES DE CLONACIÓN TAMAÑO DEL
INSERTO Vectores plasmídicos con alto número de
copias de tipo estándar 0 a 10kb
Vectores de inserción del fago lambda 0 a 10kb Vectores de reemplazamiento del fago lambda 9 a 23kb Vectores cosmídicos 30 a 44kb Bacteriófago P1 70 a 100kb Vectores PAC (cromosomas artificiales de P1) 130 a 150kb Vectores BAC (cromosomas artificiales de bacterias) hasta 300kb Vectores YAC (cromosomas artificiales de levadura) 200 a 2000kb
Capacidad de clonación de los diferentes tipos de vectores
Los más utilizados: plásmidos, bacteriófagos,cósmidos y los cromosomas artificiales de levadura
Tecnología del DNA recombinante:
Clonación: vectores y hospedadores
Plásmidos: Moléculas circulares extracromosómicas de DNAbicatenario, heredadas de manera estable durante las sucesivasgeneraciones celulares. Los más utilizados son del tipomulticopia.
(A) plásmido pBR322 (B) plásmido pUC: los plásmidospUC retiene el gen de resistencia a la ampicilina.
THR MET ILE THR ASN SER ser ser val pro gly asp pro leu glu ser thr cys arg his ala ser leu
ala LEU ALA
1 2 3 4 5 6 (1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18) 7 8
ATG ACC ATG ATT ACG AAT TCG AGC TCG GTA CCC GGG GAT CCT CTA GAG TCG ACC TGC AGG CAT GCA AGC TTG GCA CTG GCC
EcoR I Sst I Kpn ISma IXma I
BamH I Xba ISal IAcc IHinc I
Pst I Sph I Hind III
lacZ' + MCS
ori
ampr
(B)pBR322
Resistènciaa ampicilina Resistència
a tetraciclina
EcoR I Cla IHind III
BamH ISph I
Sal I
Xma III
Nru I
Ava I
Bal I
Pvu IISna I
Pst I
Pvu IRru I
ori
(A)
Tecnología del DNA recombinante
Clonación: vectores y hospedadores
Bacteriófagos Pueden actuar como vehículos naturales en latransducción de DNA bacteriano. Los fagos modificados poringeniería genética pueden hacer lo mismo a partir de cualquiertipo de DNA. Los más conocidos derivan del fago lambda de E.coli. Se dividen en vectores lambda de inserción y dereemplazamiento o sustitución.
R
R
R R R R
R R
R R
Fag de reemplaçament
Fag d'inserció
Cosmidi
Insert de 20 kb
Insert de 5 kb
Insert de 45 kb
(A)
(B)
(C)
Vectores derivados del fago lambda: (A) fago lambda de reemplazo. (B)fago lambda de inserción. (C) cósmido
Tecnología del DNA recombinante
Clonación: vectores y hospedadores
Cósmidos Plásmidos con la secuencia cos del fago lambda.Los cósmidos recombinantes son encapsula-dos en partículasfágicas con capacidad infectiva y se replican como plásmidos
en el interior de las células hospedadoras.
cósmido
Tecnología del DNA recombinante: Clonación:
vectores y hospedadores
Cromosomas artificiales. Fragmentos de DNA de granlongitud, de unas cuantas megabases: YAC, yeastartificial chromosomes.
Secuencias eucarióticas necesarias para construir un YAC:
-Dos telómeros, para la replicación completa y para laprotección de los extremos de las moléculas de DNA lineales
-Un centrómero, para la correcta segregación de lascromátidas hermanas durante la mitosis y de loscromosomas homólogos en la meiosis
-Un origen de replicación o región autónoma dereplicación (ARS), para la replicación independiente delcromosoma
También: marcador seleccionable y dianas derestricción
Tecnología del DNA recombinante
Clonación: vectores y hospedadores
Otros tipos de cromosomas artificiales que se utilizan juntocon los YAC y los cósmidos para generar mapas físicos degenomas son los BAC (“Bacterial Artificial Chromosomes”,cromosomas bacterianos artificiales) y los PAC (“Phage P1-based artificial chromosomes”, cromosomas artificialesderivados del bacteriófago P1).
Estructura de un cromosoma artificialbacteriano (BAC) utilizado para laclonación de fragmentos de DNA grandes.CMR es un marcador seleccionable deresistencia al cloramfenicol. oriS, repE,parA y parB son genes del plásmido F quecontrolan la replicación y el número decopias por célula. cosN es el lugar cos delfago lambda. HindIII y BamHI son sitiosde clonación en los que se inserta el DNAforáneo. Los dos promotores permitentranscribir el fragmento insertado. Lossitios NotI se utilizan para retirar elfragmento insertado
Tecnología del DNA recombinante
Clonación: vectores y hospedadores
Vectores de expresión
Se caracterizan por disponer de un promotor muy activo yen algunos casos regulable, que asegure la síntesis de laproteína.
El producto que se obtiene es muchasveces una proteína de fusión porque,además del producto del gen inser-tado, presenta unos pocos amino-ácidos extra en el extremo amino-terminal, propios de la proteína delhospedador
Vector de expresión de la serie pIN.
Tecnología del DNA recombinante
Clonación: vectores y hospedadores
Los hospedadores
Las bacterias son los organismos más utilizados como receptoresdel DNA recombinante, tanto por razones históricas como por lasventajas que ofrece su uso.
Con respecto a los organismos eucariotas, la levadura es uno delos más utilizados como hospedador del DNA recombinante,debido a su facilidad de manipulación en el laboratorio, su rápidocrecimiento, la abundancia de mutantes disponibles y de vectorespara su transfor-mación, y el amplio conocimiento molecular quese tiene de este organismo.
Tecnología del DNA recombinante
Genotecas: obtención y tipos
2003
Las genotecas son colecciones de clones de DNA obtenidas a partir de cierto DNA donador
Genoteca genómica: genoteca que comprende un genomacompleto
Genoteca de cDNA: genoteca compuesta de cDNA obtenidoa partir de los RNAm de un determinado tejido o tipo celular
Genoteca de expresión: genoteca realizada con un vectorque lleva señales adecuadas para provocar que cualquierinserto de DNA clonado genere un mRNA y, en últimainstancia, un producto proteico.
Tecnología del DNA recombinante
Genotecas: obtención y tipos
Método más común paraconstruir una genoteca genó-mica: fraccionamiento delDNA de partida con unaenzima de restricción y laclonación no selectiva detodos los fragmentos obteni-dos. Este método se denominamétodo de la perdigonada.
Obtención de una genoteca genómica:primero se fracciona el DNA genómico ylos fragmentos obtenidos son clonadosal azar en vectores específicos. Estosmantendrán y propagarán los respec-tivos insertos dentro de las célulashospedadoras, cada una de las cualeses portadora de un DNA recombinantediferente.
Genoma de l'organisme
Fragmentació per crear extrems cohesius o roms
Clonatge en fags o cosmidis
Amplificació i aïllament del vector en E. coli
G E N O T E C A
Tecnología del DNA recombinante
Genotecas: obtención y tipos
C C C C C C
GGG GGG
CCC AAAG G G T T T
+VECTOR
Transferasa terminal + dCTP
Oligo(dG) + DNA polimerasa (fragment Klenow)
RNAasa H
Transferasa terminal + dCTP
Oligo(dT) + transcriptasa inversa
AAA-OH 3'
TTT 5'
TTT 5'
AAA-OH 3'
TTT 5'AAA-OH
3'
TTT 5'
TTT 5'
AAACCC-OH 3'
GGG-OH 3'5'
5'
5'
3' HO-GGG
5' GGG3' HO-CCCCCC
3'
5' GGG3' HO-CCC
3' HO-CCC
3'
5'
Obtención de cDNA. Primero, se dispone deun mRNA eucariótico, portador de una colapoli(A) en el extremo 3'. La utilización deun cebador oligo(dT) posibilita la trans-cripción inversa de RNA a DNA por lapolimerasa correspondiente. Así se obtienela primera cadena de cDNA. El RNA moldese elimina con RNAsa H, y por la vía de latransferasa terminal se añaden colaspoli(dC) al extremo 3' del cDNA. Lasegunda cadena es sintetizada empleandocebadores oligo(dG) y el fragmento Klenowde la DNA polimerasa. Para facilitar laclonación del cDNA en un vector, se recurreotra vez a la transferasa terminal, queconstruye extremos coherentes entre lasdos moléculas. El DNA recombinanteformado se utiliza para transformar unacepa bacteriana
Para aislar un gen en un organismo eucariótico quizás es más fácil utilizar las genotecas construidas a partir de los mRNA, es decir, genotecas de cDNA.
Tecnología del DNA recombinante
Genotecas: obtención y tipos
Las genotecas de expre-sión requieren para suconstrucción del uso devectores de expresión loscuales se caracterizan pordisponer de todas las se-cuencias reguladoras ne-cesarias para permitir laexpresión del gen inser-tado.
Plásmidos
1
Plásmidos
2
Extremos cohesivos
Plásmido
3
Molécula de ADN recombinante
gen de resistencia a la ampicilina
Inserción de un fragmento de
clonación
ACTTTGTCCACGGCCTAAGCGTTTTTTGCCC
AGTGACTTTGTCCAAC
GTCCAACAGTTACCAAGTGACTTTGTCCAC TTTTGCCC
AGTGACTTTGTCCA ACGGCCTAAGCGTTTTTTTT
ALINEAMIENTO DE TODAS LAS SECUENCIAS Y RECONSTRUCCIÓN DEL CROMOSOMA
Secuenciación
de genomas
El método dideoxi (Sanger.1977) se basa en el empleo de didesoxinucleótidos que carecen de uno de los grupos hidroxilo, cuando uno de estos nucleótidos se incorpora a una cadena de ADN en crecimiento, está cadena no puede continuar elongándose ya que la ADN polimerasa necesita un extremo 3’ OH para añadir el siguiente nucleótido.
METODO DE SANGER (1977)
Conocimiento previo de la secuencia de ADN enfermo
Mediante ingeniería genética se construye una sonda de ADN, marcada (marcaje fluorescente), con la secuencia complementaria del ADN enfermo
ADN enfermo
ADN sano
ADN complementario del ADN enfermo
Diagnóstico de enfermedades de origen genético
ADN de la persona que se quiere diagnosticar
¿Hibridación?¿No hibridación?
Renaturalización del ADN con la sonda fluorescente
Desnaturalización del ADN
Si aparecen bandas fluorescentes demuestra que la persona presenta la anomalíaBiochip
MicroarrayDNAchip
DIAGNÓSTICO











































![[Factor Antihemofílico (Recombinante)]](https://static.fdocuments.ec/doc/165x107/62e321857857632745738462/factor-antihemoflico-recombinante.jpg)