Mitosis
description
Transcript of Mitosis
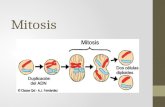
MitosisEs la divisin celular que consiste en que a partir de una clula se obtienen 2 clulas hijas, genticamente idnticas a la madre. Se produce en cualquier clula eucarionte, ya sea diploide o haploide y como mantiene invariable el nmero de cromosomas, las clulas hijas resultarn diploides, si la madre era diploide o alploide. La divisin del citoplasma se llama citocinesis, y la divisin del ncleo, cariocinesis. Algunas clulas no realizan mitosis y permanencen en un estado interfsico, pero otras la realizan frecuentemente (clulas embrionarias, clulas de zonas de crecimiento, clulas de tejidos sujetos a desgaste.). PROFASE: La cromatina se condensa para formar los cromosomas y los 2 centrolos migran a polos opuestos organizando un sistema de microtbulos (aparato mittico) para permitir la migracin de los cromosomas. 1. PROMETAFASE: Los cromosomas condensados migran hacia la placa ecuatorial del huso acromtico.2. METAFASE: Los cromosomas se alnean en el plano ecuatorial, y cada uno estn unido por su centrmero a una fibra del huso acromtico.3. ANAFASE: Las 2 cromtides de cada cromosoma se separan por fisin del centrmero y se dirigen hacia polos opuestos. El movimiento de los cromosomas hijos hacia los polos se debe a un acortamiento de las fibras cromosmicas y se alargan las fibras interzonales.4. TELOFASE: El huso mittico y los steres se desorganizan. Alrededor de cada grupo cromosmico se organiza una envoltura nuclear a partir del reticulo endoplasmtico y de la envoltura original. Los cromosomas se dispersan y retoman el aspecto de cromatina que tenan antes de iniciarse la divisin. Los nucleolos reaparecen a partir de sus organizadores.MeiosisEs un proceso de reduccin cromtica por el que los cromosomas se reducen a la mitad. En la meiosis I (etapa reduccionaria) se reduce el nmero diploide de cromosomas a la mitad (haploide) pero an los cromosomas son dobles. En la meiosis II (etapa ecuacional) se mantiene el nmero cromosmico haploide conseguido en la etapa anterior. Los cromosomas son simples. Meiosis I: Est precedida por una interfase durante la cual se duplica el materialo gentico.1. PROFASE I: La envoltura nuclear y el nucleolo se desorganizan, los centrolos migran a polos oppuestos, duplicndose y se ordena el huso acromticop. Se divide en 5 etapas: Leptonema, Cigonema, Paquinema, Diplonema y Diacinesis.2. PROMETAFASE I: Los cromosomas migran al plano ecuatorial de la clula.3. METAFASE I: Los cromosomas se alinean en el plano ecuatorial. Los 2 cromosomas del bivalente se unen por medio del centrmero a la misma fibra del uso acromtico.4. ANAFASE I: Los 2 cromosomas homlogos unidos a la misma fibra dek huso se repelen y migran a polos opuestos. Cada cromosoma est formado por 2 cromatimas.5. TELOFASE I: Cuando los cromosomas llegaron a los polos, se desorganizan el huso acromtico y los steres, se reprganizan la envoltura nuclear y los nucleolos y se constituyen los ncleos hijos.Citocinesis: Se produce simultneamentye con la telofase, y da como resultado 2 clula hijas con un nmero haploide de cromosomas. Meiosis II: Los procesos de esta divisin son semejantes a los de una mitosis en una clula haploide.1. PROFASE II: Se condensan los cromosomas, se desintegran los nucleolos, los centrolos migran a los polos y se duplican, formacin del huso acromtico y se desorganiza la envoltura nuclear.2. PROMETAFASE II: Los cromosomas condensados migran a la placa ecuatorial de la clula.3. METAFASE II: Los cromosomas se alinean en la placa ecuatorial, y cada cromosoma se une a una fibra del huso acromtico.4. ANAFASE II: Se fusiona el centrmero y se separan las 2 cromtidas de cada cromosoma. Cada una migra a un polo diferente.5. TELOFASE II: Los grupos cromosmicos llegan a los polos, el huso acromtico se desorganiza, se reorganizan la envoltura nuclear y el nucleolo, se dispersan los cromosomas y se transforman en cromatina.Citocinesis: Separacin de los citoplasmas de las clulas hijas.ecombinacin especfica de sitioLa recombinacin homloga estaba basada en la existencia de secuencias homlogas largas, por lo que la recombinacin poda producirse en diferentes sitios (resolucin en distintos lugares). La recombinacin especfica de sitio, como su propio nombre indica, ocurre en un solo lugar. Slo existe homologa de secuencia en una regin muy pequea (~10 pb), all donde se produce la recombinacin. Mecansticamente no tiene nada que ver con la recombinacin homloga, no interviene Rec A. El ejemplo ms conocido, aunque no el nico, es el de la integracin y escisin del DNA del fago lambda en el de E. coli. Los DNAs de ambos recombinan en un sitio especfico, dando lugar a un nico DNA conteniendo ambos genomas, la cepa que lo contiene se llama lisgeno, dado que eventualmente el genoma del fago puede escindirse y lisar la bacteria. Esta recombinacin es un tipo de recombinacin especializada porque ocurre entre pares especficos de secuencias cortas de DNA. Estas secuencias pueden encontrarse sobre un mismo cromosoma o en diferentes. En este caso la recombinacin no est guiada por dichas secuencias cortas, sino que depende de enzimas especializadas en reconocer dichas secuencias y catalizar cortes dentro o cerca de las secuencias y su reunin, para dar productos recombinantes. Este tipo de recombinacin conduce generalmente a un reordenamiento de la informacin.El reordenamiento gnico se promueve por la recombinacin entre dos secuencias especficas o dos copias de un elemento transponible. Si dichas secuencias se encuentran en un mismo cromosoma, se habla entonces de repeticiones (repeats) que pueden ser directas (RDs) o inversas (RIs) y el resultado de la recombinacin depende de la orientacin de las repeticiones (orientacin referida al orden de nucletidos y no a la direccin de la cadena, 3' 5' 5' 3'), como indica la figura siguiente (donde las repeticiones se representan en rojo y verde, respectivamente):
Si se trata de secuencias con orientacin inversa (RIs) el segmento de DNA que se encontraba entre las RIs queda invertido tras la recombinacin, caso (a) de la figura anterior. Por el contrario, si se trata de repeticiones directas (en la misma orientacin), despus de ocurrir la recombinacin el elemento intermedio queda delecionado como un crculo de DNA, que es el caso (b) de la figura anterior (ver tambin Fig 15.8, 15.9, Genes VII). Cuando las secuencias se ecuentran en molculas diferentes, el fragmento se integra al DNA. Como ejemplo de esta recombinacin vamos a estudiar la integracin del Fago lambda al cromosoma bacteriano. El mecanismo general se representa en la figura siguiente:
El sitio de integracin del fago se llama attP (attachment Phage), e interacciona con el sitio de integracin attB de la bacteria (attachment Bacteria). Aunque no existe homologa de secuencia entre attP y attB, existe una regin de 15 pb que es idntica en ambos, esa regin se llama O, siendo las regiones que flanquean a O, P y P' en el fago y B y B en la bacteria, as, attP es lo mismo que POP' y attB es lo mismo que BOB'. La recombinacin se da entre los sitios O de modo que al integrarse el DNA del fago en el DNA de la bacteria, el profago (DNA de fago integrado) quedar flanqueado por dos nuevos sitios: BOP' y POB'. BOP' es lo mismo que attL (a la izquierda del profago) y POB' es lo mismo que attR (a la derecha del profago).
Debido a que el DNA del fago es circular, el evento recombinacional permite su insercin en el DNA bacteriano como una secuencia lineal. Esta forma integrada se denomina profago y queda flanqueada a la izquierda por attL que consiste en (BOP') y a la derecha por attR (POB'). Estos sitios attL y R estn involucrados en la escisin del profago que transcurre por el mecanismo opuesto al de la integracin. Ello implica que los eventos de recombinacin para lograr la integracin y la escisin son "reversibles" aunque se dan en condiciones diferentes.El conjunto de protenas que participan en uno y otro porceso se resumen en la siguiente tabla:ReaccinRecombinantesGenes y enzimas que participan
Fago lambdaBacteria
IntegracinAttB x AttPIntIHF (Factor de integracin del hospedero), genes himA y himD1
EscisinAttL x AttRXis, IntIHF
1 Es una protena de 20 kD, no esencial para la E. coli y no requerida para la recombinacin homloga bacterial. Tiene la habilidad de enrollar DNA en su superficie.La reaccin de integracin se produce en condiciones normales porque el fago es un fago temperado, es decir que tiende a la lisogenia, a integrarse en el genoma y mantenerse latente. La recombinacin especfica de sitio puede ser llevada a cabo in vitro por int e IHF. Involucra la rotura y reunin precisa en ausencia de cualquier sntesis de DNA. Tanto los sitios como las protenas que intervienen en la integracin y la escisin son diferentes, por lo que son dos procesos distintos, y no uno reversible. La protena int, tiene las actividades tpicas de una recombinacin, endonucleasa y ligasa. Introduce dos cortes no enfrentados, 7 pb, tanto en attP como en attB y se unen unos extremos con otros, la secuencia tiene que ser la misma para que se apareen. El mecanismo de la reaccin es similar a las topoisomerasas y la protena A de X164, la protena int corta el DNA dejando un grupo 5' OH libre, quedando de forma transitoria unida covalentemente por un residuo de tirosina al extremo 3' y a continuacin se ligan los extremos del DNA. En la reaccin intervienen cuatro molculas de int, dos en cada DNA. Dos protenas int cortan y ligan, dando lugar a un intermedio de Holliday. Realmente no es un verdadero intermedio de Holliday, ya que el punto de cruce no puede desplazarse, lo nico que hay en comn en esta regin es una zona de 15 pares de bases y por tanto el intermedio es esttico en lugar de dinmico. Este intermedio lo resuelven las otras dos molculas de int, actuando de la misma manera, es decir, cortando y uniendo. El mecanismo puede representarse como sigue (los crculos grises representan subunidades de int):
La reaccin de integracin es estereoespecfica, es decir, el DNA tiene que tener una conformacin determinada que le da IHF. IHF (Integration Host Factor = Factor de Integracin del Hospedador) no tiene actividad cataltica sino un papel arquitectnico, hace que se doble el DNA de un modo determinado para que la reaccin se lleve a cabo. Aunque IHF se descubri en la integracin del fago lambda, interviene en muchos otros procesos que no estn relacionados. IHF forma dmeros, reconoce especficamente a una secuencia y dobla el DNA unos 140. Se conoce la estructura terciaria del dmero de IHF unido al DNA, el siguiente modelo muestra una porcin de attL al cual se ha asociado un dmero IHF y provocado una curvatura que acerca los sitios de recombinacin C' y P1' a int (en verde), el punto de clivaje se indica con una flecha roja:
Al doblarse el DNA se acercan dos sitios de interaccin con int, IHF dobla el DNA de dos maneras (1) desde abajo, interaccionando con los grupos fosfato del DNA o (2) desde arriba, mediante lminas con residuos de prolina que actan a modo de cua intercalndose entre dos bases.En la figura siguiente se muestran los sitios a los que se une la protena int (crculos), hay siete sitios de interaccin en POP' y otros dos en BOB':
IHF se une a tres sitios (cuadrados). Al interaccionar los tres IHF el DNA adopta una conformacin determinada. Todo esto forma un complejo nucleoprotico llamado intasoma, donde la protena int lleva a cabo la rotura y unin, y la IHF dobla el DNA para que pueda darse la reaccin. Otra representacin muestra la conformacin del DNA en el sitio POP, attP es mucho ms grande que attB:
IHF se une a cada uno de los tres sitios de unin en attP provocando en cada caso una curvatura de 140 de manera qeu el DNA queda en una conformacin de espiral. La funcin de attP requiere 240 pb, pero la de attB slo 23 pb, en la cual hay solo 4 pb sobre cada lado del centro. La diferencia de tamao entre ellos sugiere que juegan diferentes papeles en la recombinacin. La Fig 14.21, Genes VII muestra que si attP y attB sufren los mismos cortes escalonados, extremos simple cadena pueden quedar disponibles para cruzamientos. La distancia entre los puntos de entrecruzamiento es de 7 pb y la reaccin genera extremos 3'fosfato y 5'OH.La reaccin in vitro requiere superenrrollamiento en attP, pero no en attB. Cuando la reaccin se lleva a cabo in vitro entre dos molculas de DNA se, casi todo el superenrrollamiento es retenido por los productos. Quiere decir, que no hay intermediarios libres en los cuales ocurra rotacin. Esto es consistente con la idea que la reaccin procede a travs de una unin Holliday. La ruptura y reunin se parece a la actividad de topo I.Recombinacin homloga en E.coliAl menos 25 tipos distintos de protenas involucradas en recombinacin homloga en E.coliRecA, RecBCD, RecF, RecG, Re cJ, RecN, RecO, RecQ, RecR, RuvAB, RuvC, PriA, SSB, DNA polimerasas, DNA topoisomerasas y DNA ligasas.
Secuencia en cis (hotspot de recombinacin)GCTGGTGG Genoma de E.coli 1009 sitios (1/6kb)
Recombinacin Homloga Ruptura de la doble hlice en 2 molculas de DNA homlogas y unin cruzada Sitio de intercambio secuencias homlogas Heteroduplex en sitio de intercambio No hay alteracin de la secuencia en el sitio de intercambio
Sistema RecBCD (E.coli)Esencial para el 99% de eventos de recombinacin en DSB en E.coliIniciacin Procesamiento del DSB (RecBCD y RecQ) Formacin del filamento infectivo (RecA)Apareamiento de homlogos e intercambio de cadenas Invasin de la doble hliceExtensin del heteroduplex Complejo de protenas motoras RuvABResolucin Endonucleasa RuvC (parte del complejo RuvAB)RECOMBINACIN SITIO ESPECFICAReaccin entre dos sitios especficosLargo de sitios blanco 14-50pbPueden ser secuencias homlogas o noEnzimas involucradas especficas (recombinasas)Integracin del Fago lambda (Recombinasa= Int)Fago P1 (Recombinasa Cre / sitios loxP)Resolucin de cointegrados (plsmidos multimricos) Sistema de recombinacin sitio especfica Xer (Cromosoma Bacteriano). En E.coli: sitio blanco (dif, 28pb) + 2 recombinasas XerC y XerD