La importancia de la masa exacta: Identificación de ... · British American Tobacco, UK Mass...
-
Upload
truongdiep -
Category
Documents
-
view
225 -
download
0
Transcript of La importancia de la masa exacta: Identificación de ... · British American Tobacco, UK Mass...
La importancia de la masa exacta:
Identificación de desconocidos y
análisis diferencial de muestras
Jaume C. Morales
Especialistas de Producto
Agilent Technologies Spain Tarragona / Lleida Octubre de 2012
• Existe in creciente uso de las ciencias ómicas
• Las técnicas usadas són : Cromatografía más espectrometría de masas en
combinación con análisis estadístico.
• Las areas donde las ciencias ómicas están incrementando su presencia son :
– Salud y enfermedad (clinica), nutrición, calidad alimentaria
– Control de calidad en Fermentación
– Biofueles “green technologies” e investigación en cereales
– Monitorización ambiental
• Aspectos críticos en el flujo de trabajo:
– Preparación de muestra y automatización
– Identificación de los compuestos
– Integración de los resultados quimiométricos en contexto biológico (biociencia)
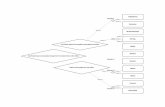
Ciencias ómicas
Agilent ….omics Workflow G
CM
S
LC
MS
Separate &
Detect
GC/MSD
GC-QQQ
LC-TOF/QTOF
LC-QQQ
Feature
Finding
Quantitate
MassHunter Qual AMDIS or Find by
chromatographic
deconvolution
MassHunter Qual MFE,
Find by Formula,
Find by Ion
Alignment &
Statistics
Mass Profiler
Professional
(MPP)
Identify
ID Browser
Pathways
Pathway module
Cytoscape
Mass Profiler
(MP)
Emerging Insights:
Metabolomics at Agilent 5990-6048EN
hydrophilic
hydrophobic
ionic
uncharged
Phospho sugars
amino acids
nucleotides
lipids
organic acids
amines
TCA cycle
pentose-phosphate path
glycolysis purine metabolism
proteins
peptides
sugars
hydrocarbons
phospholipids
quinones
aminoglycosides
cell membrane cell wall
amino acid metabolism
Métodos de Separación
Sulphated sugars
Análisis dirigido o indiscriminado
Adquisición Indiscriminada (untargeted adquisition) – TOF / QTOF
• Perfil de todos los compuestos detectables
• Significación de los datos: Basado en un enfoque de descubrimiento.
• Caracterización de los compuestos detectados por : m/z, abundancia, espectro y tiempo ret.
• Los compuestos se identifican despues del analisis diferencial
• La Identificación puede ser dirigida por búsqueda en bases de datos concretas, (metabolitos, pesticidas, compuestos toxicologicos, etc…) e incluso localizarlos en rutas metabólicas (biociencia)
Adquisición Dirigida (targeted acquisition) – QQQ
• Sólo de compuestos conocidos
• Mayor sensibilidad que el enfoque Indiscrimando
• Cuantificación absoluta – Necesita patrones externos e internos
• Desarrollo de metodos con SIM o MRM para cientos de compuestos (bases de datos)
• Susceptible de analisis diferencial para validación.
El análisis Indiscriminado puede utilizarse para
identificación Discriminada
La adquisición de datos Indiscriminada
se efectúa guardando el espectro total
en cada punto del cromatograma
La información es útil para usos a posteriori.
Los compuestos que el análisis estadístico
revela como diferenciales pueden dar un
significado al grueso de los datos originales.
El Software permite convertir puros datos
en una interpretación significativa.
El listado de compuestos diferenciales
puede enviarse de nuevo al equipo para
que realice ahora una Adquisición Dirigida.
Mass Hunter Targeted Data Mining For LC/MS:
Find by Formula (FBF)
Se trata de una búsqueda dirigida
usando una base de datos de masa
exacta de los compuestos de
nuestro interés:
• Calcula los iones basándose en las
fórmulas empíricas que le
proporcionamos
• La coincidencia de Retention time es
opcional
• Trabaja sobre bases de datos Agilent
PCDL o genéricas .xls, .csv
• El resultado de la búsqueda puede
revisarse en Mass Hunter Qual
• Produce un registro .cef para
exportar a Mass Profiler Professional
Compound chromatogram
Compound
spectra
MassHunter PCDL Manager B.04.00 Cree y edite su propia Base de datos de compuestos con masa
exacta, Tr y espectros MS/MS
Personal Compound Database and Library Manager (PCDL)
Añada compuestos con su información de Masa exacta, RT, espectro
MS/MS demasa excata a multiples EC, enlaces con otras web’s
Realize rápidas busquedas manuales interactivas.
Realize rápidas busquedas automáticas de lotes de compuestos.
Uso directo o incluido en Mass Hunter.
James Hutton, UK
Institute Heidger, Germany
University of Tuebingen, Germany
British American Tobacco, UK
Irish Distillers, Ireland
MARS, UK
Casos Reales
James Hutton Institute , UK
Se analizaron dos extractos de patata (SGT y D) en un QTOF de alta resolución en
modos MS y Auto MS/MS.
Los datos se procesaron a través del algoritmo de deconvolución con masa exacta
MFE (Molecular Feature Extractor) buscando todos los compuestos ionizables en las
muestras y el resultado se inspeccionó sobre la base de datos METLIN. Para
aquellos compuestos detectados pero AUSENTES en METLIN se pidió al software
que propusiera la formula elemental más probable a través del algoritmo MFG
(Molecular Formula generation )
Adicionalmente se exportó en un registro .CEF todos los compuestos detectados en
todas las muestras y comparadas con Mass Profiler y Mass Profiler Professional.
El enfoque “metabolómico” usado aquí usó solamente 5 replicas técnicas (n=5) con
el posible sesgo que eso pueda provocar en los datos.
James Hutton Institute , UK / Resultados
Este ejemplo nos muestra datos del análisis de la muestra D. Se detectaron 1895 compuestos con una
abundancia mayor que 2500. El cromatograma muestra la superposición de los EIC de la m/z de esos
compuestos junto al TIC (negro) así como una ampliación en la zona 6,5 – 9,7 min para observar mejor el
detalle.
Mass Profiler Professional: suministra un paquete de análisis estadístico más sofisticado. Eso permite la
comparación de múltiples conjuntos de muestras. En éste ejemplo se comparan 3346 compuestos a través
de las 10 muestras (5 por tipo de patata).
James Hutton Institute , UK / Resultados
Mass Profiler Professional : La construcción del análisis estadístico puede hacerse a través de un Tutorial (Guided Workflow) o
en modo avanzado (Advanced Workflow) para usuarios expertos. En dicha construcción los datos se filtran, alinean y
normalizan. A posteriori el Tutorial permite una serie de filtros que nos reduce el número de artefactos. Aquí se muestra como
sólo 2302 “entities” de 3346 están presentes en al menos 2 muestras.
James Hutton Institute , UK / Resultados
Mass Profiler Professional : Aquí se muestra una mayor reducción de “entities” pasando el filtro tan sólo
aquellas que aparecen en el 100% de todas las muestras que pertenecen a una condición (tipo de patata).
James Hutton Institute , UK / Resultados
Mass Profiler Professional : Como parte del Tutorial se ejecuta un análisis por componentes principales (PCA) que nos permite
hacer un QC de las muestras en cuanto a representatividad , uniformidad y diferenciación. Esto nos permite comprobar si existen
compuestos que realmente diferencian las dos condiciones. En éste caso las bolas rojas representan a las Patatas D y las azules
a las Patatas SGT.
James Hutton Institute , UK / Resultados
Mass Profiler Professional : Finalmente se realiza el análisis estadístico (t-Test) y se muestra en forma de “Volcano plot” de
SGT vs Desire. Esto nos muestra 476 compuestos cuyo fold change es >2.0 ( Log [cambio abundancia]) y el valor de
(probabilidad) P es <0.001. Los compuestos (cuadros rojos) en el panel de la derecha aumentaron en SGT respecto a Desire
mientras que los del panel de la izquierda decrecieron.
James Hutton Institute , UK / Resultados
Mass Profiler Professional: En éste gráfico cada linea representa a un compuesto y su abundancia en Patatas Desire y SGT.
Cada uno de éstos compuestos viene caracterizado por su Rt y masa exacta.
James Hutton Institute , UK / Resultados
Mass Profiler Professional: El menú lateral de Workflow permite a partir del resultado del análisis diferencial tomar acciones.
En éste caso se realiza un ID Browser que nos permite identificar los compuestos aquí mostrados en una base de datos de
masa exacta y/o generar fórmulas moleculares empíricas.
James Hutton Institute , UK / Resultados
Mass Profiler Professional:. Para éste ejemplo se han volcado 476 para identificar por ID Browser. La Base de Datos
utilizada para identificar ha sido METLIN (>23.000 entradas). La búsqueda se ha realizado solamente en base a la masa exacta
aunque se podría incluir también el Tiempo de retención.
James Hutton Institute , UK / Resultados
Mass Profiler Professional: De cara a tener una mayor confirmación de algunos compuestos se puede exportar la lista de
éstos a una lista de inclusión para QTOF en modo dirigido pues conocemos ya su m/z y Tiempo de retención. Esta lista se
importa directamente desde MH Acquisition y se inyectan de nuevo las muestras. Los datos resultantes con espectros MS/MS
pueden buscarse en librerías de espectros MS/MS . La librería Agilent METLIN incluye >6000 espectros MS/MS.
James Hutton Institute , UK / Resultados
• Dos extractos de diferente patata se han analizado por LC/MS y se han detectado los
compuestos presentes por MFE
• Usando Mass Profiler Professional, se observaron diferencias significativas entre éstas
muestras.
• De >3000 entities, 467 mostraron ser significativamente diferentes en términos de
abundancia entre las dos muestras.
• Las entities significativas pueden ser Identificadas con MPP usando Bases de Datos y/o
Generando fórmulas Moleculares empíricas.
• La lista de entites significativa puede exportarse como lista de un inclusión para hacer un
Análisis de adquisición Dirigido por MS/MS y buscar sus espectros en librerías
espectrales MS/MS
James Hutton Institute , UK / RESUMEN
Institute Heidger, Germany
Comparación de muestras de Vino y Whisky usando Mass Profiler Professional: PCA Analysis.
Institute Heidger, Germany
Comparación de muestras de Vino y Whisky usando Mass Profiler Professional: Predicción de Modelos
University of Tuebingen, Germany
En este ejemplo Mass Profiler se utilizó para evaluar las diferencias entre dos muestras de agua 2112 y 2012
para detectar el origen de una contaminación en un rio. Los gráficos muestran la Masa vs. Tiempo de
Retención de los compuestos encontrados por MFE . Se aprecia una marcado incremento de compuestos
encontrados en la Muestra 2012.
Sample 2112
Sample 2012
Una comparación estadística usando 5 réplicados (n=5) permite la visualización de compuestos únicos de
cada muestra. Los gráficos muestran aquellos compuestos que sólo estan en una de las dos muestras.
De un total de 766 compuestos encontrados :
579 estaban sólo presentes en las muestras 2012
33 compuestos sólo en las muestars 2112.
El resto estaban en ambas muestras
University of Tuebingen, Germany / Results
British American Tobacco, UK
Mass Profiler tiene la capacidad de compara dos conjuntos de muestras. Primero se extraen los compuestos
por MFE y luego se alinean y comparan. Al igual que MPP, los compuestos significativamente diferentes (t-
Test solo) pueden identificarse por Bases de Datos o Generación de Fórmula. En el gráfico inferior puede
observarse una comparación entre las muestras 1 y 4 . El gráfico muestra sólo aquellos compuestos que han
aumentado su abundancia en la Muestra 4 respecto a la Muestra 1.
Cada uno de los compuestos puede estudiarse para determinar su distribución de abundancia. P.e. El compuesto nº 9 abajo.
British American Tobacco, UK / Results
Aquí se muestra un listado de compuestos identificados por ID Browser. Algunos a través de base de datos y otros generando
fórmulas.
British American Tobacco, UK / Results
4. Results
Expandiendo cada compuesto nos muestra los diferentes resultados de la búsqueda con el Overal Score donde por defecto
es seleccionado el máximo. The BEST.
Mass Profiler al igual que MPP puede tambien generar listados de inclusión para adquisición MS/MS en QTOF dirigido.
British American Tobacco, UK / Results
Análisis Quimiométrico
Mass Profiler y Mass Profiler Professional proporcionan potentes herramientas
estadísticas para el análisis diferencial.
Este enfoque puede aplicarse a cualquier estudio de cambios en un proceso con datos
MS.
Los resultados de MP y MPP pueden exportarse a una lista de inclusión para análisis
MS/MS dirigido.
La lista de inclusión se importa directamente desde MH Acquisition
Pathway analysis es único de Agilent (Biociencia)
Agilent proporciona bases de datos de masa exacta :
METLIN PCD(L) unica de Agilent
Pesticidas
Compuestos Toxicológicos
Food Authenticity Determinations:
Metabolomic profiling of wines using
HPLC-QTOFMS coupled to Agilent's advanced
data mining and statistical tools
Ondrej Lacinaa, Lukas Vaclavika, Jerry Zweigenbaumb
a Institute of Chemical Technology Prague, Czech Republic b Agilent Technologies, Wilmington, DE, USA
EXPERIMENTAL SETUP
INSTRUMENTAL ANALYSIS
• RRLC separation, RP column
• ESI(MM)-QTOFMS (MS and MS/MS)
DATA MINING
• molecular feature extraction
• background subtraction
DATA PROCESING • data filtering
• statistical analysis (ANOVA, PCA)
• classification model (PLS-DA)
IDENTIFICATION
• MS/MS experiments
• molecular formula estimation
• database search
SAMPLES
• Red wines (45 samples total)
• Varieties: Cabernet Sauvignon (15), Merlot (16), Pinot Noir (14)
• Countries of origin: Czech Republic, Slovakia, France, Italy,
Macedonia, Bulgaria, Hungary, Australia, Chile, Germany, USA
• Vintage: 2004 - 2008
VERY VARIABLE SAMPLE SET…
SAMPLES
INSTRUMENTATION
Agilent Technologies 6530 Accurate-Mass Q-TOF LC/MS
Agilent Technologies 1200 RRLC system
Jet Stream ESI source
NO SAMPLE PREPARATION, DIRECT ANALYSIS OF SAMPLE…
Multimode ion source
Eclipse Plus C18 (2.1×100, 1.8µm)
HILIC Plus C18 (2.1×100, 3.5µm)
TIC of wine and LC blank, LC-(ESI+) QTOFMS
?
? ?
? ?
? ? ? ?
? ? ?
? ?
? ?
?
? ? ? ?
? ?
?
?
? ?
? ?
?
? ?
? ?
?
?
DATA MINING
BLANK WINE
Complex data, only valid
compounds needed to be
extracted.
Settings of extraction method
Background ions to
be subtracted
DATA MINING: FIND BY MOLECULAR FEATURE
DATA PROCESSING
Agilent Technologies Mass Profiler Professional Software
…is often very tedious and time demanding with metabolomic based studies…
DATA MANAGEMENT DATA FILTERING STATISTICAL TOOLS DATA INTERPRETATION
DATA PROCESSING
Aligning MFs (entities) across the data set
20506 entities
obtained from all
samples
DATA PROCESSING
Filtration of entries by frequency (occurrence in classes)
Default filter setting: presence of
an entity in 100% of samples at
least in one group.
In very variable sample sets, some significant compounds
could be filtered out…
Ave
rag
e i
nte
nsit
y
663 entities from
20506 remained
DATA PROCESSING
Filtration by frequency : Example of significant entity for
Pinot Noir
This compound is
present in 14 of
15 Pinot Noir
samples
Only few significant entities are present in 100% samples in a class
Ave
rag
e i
nte
ns
ity
DATA PROCESSING
3600 entities from
20506 remained
Change the default
value to 50%.
Filtration by frequency
Ave
rag
e i
nte
nsit
y
DATA PROCESSING
Statistical analysis: Fold Change
Fold Change Analysis is used to identify entities
with significantly different abundance in selected
classes.
Cabernet Sauvignon and Merlot was a
problematic pair, cut-off FC ≥ 2.0 was applied…
Ave
rag
e i
nte
nsit
y
26 entities from
40 remained
DATA PROCESSING REVEALING THE INTERNAL STRUCTURE OF THE DATA USING PCA…
PCA of the data initial entries (20506)
PCA of the data entries filtered by frequency (3600)
CABERNET SAUVIGNON
MERLOT
PINOT NOIR
FILTRATION
DATA PROCESSING REVEALING THE INTERNAL STRUCTURE OF THE DATA USING PCA…
PCA of the data Entries after ANOVA (p≤0.05) & Fold Change (≥2.0)
(26)
CABERNET SAUVIGNON
MERLOT
PINOT NOIR
FILTRATION
DATA PROCESSING BUILDING CLASS PREDICTION MODEL
Mass Profiler Pro enables use of many methods for construction of class prediction models…
DECISION TREE
ARTIFICIAL NEURAL NETWORKS
SUPPORT VECTOR MACHINE
NAIVE BAYESIAN
PARTIAL LEAST SQUARE DISCRIMINATION
DATA PROCESSING BUILDING CLASS PREDICTION MODEL
Mass Profiler Pro enables use of many methods for construction of class prediction models…
DECISION TREE
ARTIFICIAL NEURAL NETWORKS
SUPPORT VECTOR MACHINE
NAIVE BAYESIAN
PARTIAL LEAST SQUARE DISCRIMINATION
DATA PROCESSING BUILDING CLASS PREDICTION MODEL
MODEL TRAINING
MODEL VALIDATION
• Calculation of discriminant functions from actual input data set
• Model is applied to input data set
• Classes in input data set are randomly divided into N equal parts, N-1 parts
are used for model training, remaining part is classified with the use of
obtained model
• The whole process is repeated N-times
MODEL RECOGNITION ABILITY
MODEL PREDICTION ABILITY
DATA PROCESSING
CONFUSION MATRIX - VALIDATION
• During model validation , two MERLOT samples were misclassified…
• All Cabernet Sauvignon and Pinot Noir samples were classified correctly
• The prediction ability of the model is 95.6%
Agilent 7200 GC/Q-TOF Applications.
The Experience so far
Sofia Aronova GC/Q-TOF Application Chemist
Agilent Technologies, Santa Clara
Food Testing and Flavors:
Caracterización del aceite de
oliva
• Procesado estadístico con MPP para los datos del GC/Q-TOF
• Búsqueda de espectros MS/IE de los espectros del GC/Q-TOF
• La ionización química proporciona información de masa exacta del
ión molecular
1. Las muestras de aceite de oliva fueron sometidas a un test sensorial y clasificadas en
PASA o FALLA
2. Adquisición por GC/Q-TOF con ambas fuentes IE y IC
3. MassHunetr Qual se utilizó para la deconvolución y listado de compuestos
encontrados que se volcó como registro CEF a MPP para efectuar el análisis
estadístico.
4. MPP después del análisis estadístico, se utilizó para la construcción del modelo de
predicción.
5. El modelo fue capaz de predecir correctamente si las muestras pasarian el test
sensorial o no.
Caracterización del aceite de oliva : Estrategia
Objetivos:
• Crear un modelo que pueda predecir si una muestra pasaría o no el test
sensorial
• Identificar compuestos en el aceite estadísticamente significativos presentes
a diferentes niveles dependiendo si pasó o no el test
Caracterización del aceite de oliva : Filtrado de datos
La deconvolución
cromatográfica encontró 442
compuestos únicos, la mayor
parte de los cuales se
manifestaron tan sólo en una
ocasión o dos y por lo tanto
fueron descartadas por MPP.
La tabla lista cuantos de esos 442 compuestos
aparecían en cada muestra.
Caracterización del aceite de oliva : Visualización del
agrupamiento de datos (Data Clustering)
Principal Component Analysis (PCA) del MPP ayuda a visualizar el
agrupamiento de datos.
falla
pasa
Caracterización del aceite de oliva : Fold Change Analysis
El Volcano Plot (derecha) muestra el “Fold Change” para cada “entity” en el eje de
X y su significancia en el eje de Y.
Compuestos presentes
en las muestras que
fallaron el test sensorial.
Caracterización del aceite de oliva : Búsqueda en Librerias
La librerías comerciales de IE aún siendo de masa unitaria puede utilizarse para
comparación por espectros usando datos de masa exacta de IE GC/Q-TOF para
identificar compuestos
Espectro del compuesto
Espectro en librería NIST
Espectro del compuesto
(masa exacta)
EI
Caracterización del aceite de oliva : Resultados MPP
• El modelo predijo correctamente las muestras incluyendo aquellas no
utilizadas para construir el modelo.
• Las muestras NO utilizadas para la creación del modelo se listan bajo el
parámetro “Training” como ‘None’