Genética replicación, transcripción y traducción
-
Upload
asancheztome1550 -
Category
Documents
-
view
19.651 -
download
11
Transcript of Genética replicación, transcripción y traducción

El ADN como material genéticoA comienzos del siglo XX, se había demostrado que los genes se localizan en los
cromosomas. Dada la composición nucleoproteica de estas estructuras, los ácidos nucleicos y las proteínas eran las sustancias candidatas a constituir los genes.
En 1928 Frederick Griffith trabajando con la bacteria que provoca neumonía en los humanos (Streptococcus pneumoniae) y que resulta letal para los ratones, demostró que la capacidad biológica para producir una cápsula (hecho que determina la virulencia de las bacterias) podía ser adquirida de otra cepa por medio de una sustancia, aún no identificada, a la que denominó “factor transformante”. Griffith había observado que existían dos cepas distintas, unas virulentas, que provocan la muerte, provistas de una cápsula de polisacáridos que da a sus colonias un aspecto liso, por lo que las denomino cepas S, y otras aparecidas por mutaciones de las anteriores, que son inofensivas, y que carecen de esa cápsula de polisacáridos, motivo por el cual sus colonias no tienen aspecto liso sino rugoso, a las que denominó cepa R. La cápsula de polisacáridos forma una capa mucosa que protege a las bacterias de los fagocitos.
Al parecer, las mutantes de la cepa R carecen de alguna enzima necesaria para sintetizar dicha cápsula, por lo que son fagocitadas. Griffith inoculó a los ratones bacterias S muertas por ebullición y observó que éstos sobrevivían, con lo que demostró que las cápsulas no eran las que provocaban la enfermedad. Al infectar un ratón con una mezcla de bacterias S (virulentas) muertas y de bacterias R (no virulentas) vivas, el ratón inexplicablemente enfermaba y moría. Además en su organismo podían aislarse bacterias S vivas. Algo había en los cadáveres de las bacterias S que había provocado la transformación de las R en S.
En 1944, Avery, McLeod y McCarthy intentaron aislar y determinar la molécula que había hecho que los neumococos R se transformaran en S en los experimentos de Griffith; demostraron experimentalmente que sólo los extractos de bacterias S muertas que contenían ADN eran capaces de producir dicha transformación; luego el ADN era la molécula portadora de la información biológica, la que suministraba la información de cómo se sintetizaba la cápsula de polisacáridos, que hace a estas bacterias resistentes a los fagocitos.
Cultivaron las bacterias en tubos de ensayo; fueron destruyendo sistemáticamente, uno por uno, todos los componentes bioquímicos de las células S, para evitar la transformación e identificar a su responsable.
1
1

Degradaron los polisacáridos y la transformación seguía produciéndose; por tanto, dedujeron que la cubierta no era el principio transformante. A continuación, destruyeron las proteínas de las células S y vieron que seguía produciéndose la transformación. Lo mismo ocurrió cuando destruyeron el ARN. Por fin, expusieron los extractos de células S a la enzima destructora de ADN (la ADNasa) y no se produjo la transformación: el principio transformante era el ADN.
A pesar de la evidencia de este experimento, el mudo científico se resistió a acaptar que el ADN era el portador de la información biológica. Muchos consideraron que se trataba de una peculiaridad de las bacterias. No se entendía cómo una molécula constituida por sólo cuatro tipos de nucleótidos podía ser la que contenía la información biológica, en vez de las moléculas proteicas que están constituidas por 20 tipos de aminoácidos. Fue necesario el experimento de A. Hershey y de M. Chase en 1952 para confirmar estas conclusiones.Hershey y Chase trabajaron con un virus de ADN que infectaba a las bacterias, el bacteriófago T2. A partir de un cultivo con el isótopo radiactivo S35 como fuente de azufre, consiguieron virus que tenían este isótopo en los aminoácidos (cisteína y metionina) de su cápsida proteica, y a partir de otro cultivo con el isótopo radiactivo P32 consiguieron obtener virus que tenían este isótopo en su ADN. Posteriormente infectaron con estos virus un cultivo de la bacteria Escherichia coli. Tras dejar tiempo suficiente para que los virus infectaran a las bacterias, agitaron el cultivo, mediante una batidora doméstica, para separar las cápsidas vacías de los virus infectantes, y finalmente las retiraron mediante centrifugación. A partir del primer cultivo observaron que la radiactividad debida al S35 quedaba en las cápsidas vacías, mientras que con el segundo cultivo constataron que el ADN radiactivo aparecía en el interior de las bacterias y, al cabo de tiempo, en el interior de los nuevos virus descendientes. Así se demostró que era el ADN, y no las proteínas, la molécula que pasa de una generación a otra y, por lo tanto, la que debe contener la información de cómo han de ser los organismos.
Material genético en procariotas y eucariotasMendel utilizó el término “factor hereditario” (actualmente sustituido por el
término gen) para referirse a los factores responsables de cada carácter. Demostró que cada carácter está determinado por dos factores hereditarios.
Antes de 1940 se consideraba que el gen era la unidad de información hereditaria, o sea, que el gen era la unidad más pequeña capaz de controlar un carácter.
En 1941 Beadle y Tatum enunciaron la hipótesis denominada “un gen-una enzima”, según la cual cada gen (fragmento de ADN) lleva información para una enzima. Según esta hipótesis, un gen contiene la información para que los aminoácidos se unan en un determinado orden y formen una enzima. La forma como un gen controla un carácter es mediante la producción de una enzima que actúa en una determinada ruta metabólica, posibilitando la síntesis de una determinada sustancia.
Con posterioridad, esta hipótesis fue ampliada, ya que el gen podía codificar una proteína cualquiera, no necesariamente enzimática, aunque estas últimas, al permitir las reacciones metabólicas, tienen efectos fisiológicos más notables. Por otra parte, como algunas proteínas están constituidas por más de una cadena polipeptídica, resultaba más apropiado decir que cada gen codifica solamente una cadena polipeptídica. El gen es una unidad de transcripción.
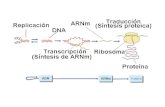
Quedaba claro que la expresión del mensaje genético, es decir, la ejecución de las órdenes contenidas en la molécula de ADN, consiste en la síntesis de proteínas específicas. Sin embargo, dado que la síntesis de proteínas se realiza en los ribosomas (situados en el citoplasma) y que el ADN se encuentra en el núcleo, del que no sale, se hace necesaria la existencia de alguna molécula que actúe como intermediario entre el ADN y los ribosomas. Este papel de intermediario lo realiza un tipo de ARN, el ARN mensajero (ARNm). El proceso de formación del ARN se denomina transcripción.
2
2

Con la información contenida en la molécula de ARNm se puede sintetizar una cadena polipeptídica en un proceso denominado traducción que ocurre en los ribosomas. En este proceso intervienen otros tipos de ARN, el ARN ribosómico (ARN r), componente fundamental de los ribosomas, y el ARN de transferencia (ARNt), que transporta los aminoácidos hasta los ribosomas.
En 1970 Francis Crick enuncia el dogma central de la Biología molecular :
El ADN forma una copia de parte de su mensaje sintetizando una molécula de ARN mensajero (proceso denominado transcripción)(se realiza en el núcleo), la cual constituye la información utilizada por los ribosomas para la síntesis de una proteína (proceso de traducción).
Todo este trasvase de información se produce gracias a la naturaleza química de los ácidos nucleicos. Debido a la complementariedad de las bases nitrogenadas, el ADN se puede replicar y transcribir a ARNm, que se va a traducir por medio del ARNt y ARNr, también gracias a la complementariedad de las bases.
En la actualidad el dogma ha tenido que ser modificado, debido a los mecanismos de replicación de algunos virus:
1. Algunos virus que almacenan su información genética en forma de ARN poseen una enzima, la ARN replicasa, capaz de fabricar copias de este ARN.
2. Los retrovirus almacenan su información genética en una molécula de ARN. Emplean una enzima, la transcriptasa inversa, que sinteriza ADN a partir de una molécula de ARN. El proceso recibe el nombre de retrotranscripción o transcripción inversa.
Estructura del genomaEl genoma de un organismo es su material genético, es decir, su ADN.La secuencia de bases nitrogenada del ADN constituye la información codificada
que las células emplean para sintetizar sus proteínas. Sin embargo la organización de esa secuencia es distinta en las células procariotas y en las eucariotas
En las células procariotas prácticamente todo el ADN se emplea como información para la síntesis de proteínas y el gen codificador da cada proteína se compone de una secuencia continua de nucleótidos (carece de intrones). El ADN contiene una copia de cada gen. No hay apenas ADN silencioso (que no se transcribe). Su genoma está formado por un solo cromosoma circular. Además de este ADN cromosómico, muchas bacterias contienen plásmidos, que son moléculas de ADN circulares más pequeñas que el cromosoma y con capacidad para replicarse independientemente de el.
3
3

Organismos eucariontes: La mayor parte de su ADN se encuentra en el núcleo, constituyendo el genoma nuclear. No existe una relación directa entre la complejidad del organismo y la cantidad de ADN, ya que la mayor parte de este no codifica proteínas ni interviene en la regulación de su síntesis.
En las células eucariotas parece existir un exceso de ADN, ya que solamente un 10% o menos de esa molécula se emplea para codificar proteínas. El resto tiene funciones poco conocidas. Se estima que en la especie humana menos del 5% del ADN codifica proteínas.
Un importante porcentaje de este ADN no codificante está formado por secuencias de nucleótidos altamente repetidas. Casi la mitad del ADN en eucariotas es altamente repetitivo, lo que significa que existen secuencias de nucleótidos repetidas cientos o miles de veces. El ADN altamente repetitivo no lleva información para la síntesis proteica y quizá desempeña un papel importante en la estabilidad de la estructura de los cromosomas.
Otra característica distintiva del material genético de las células eucariotas es que las secuencias nucleotídicas que codifican para proteínas no suelen ser continuas, sino que existen secuencias no codificadas intercaladas. Los fragmentos de ADN codificadores se denominan exones, mientras que los que no llevan información para la síntesis proteica se llaman intrones.
Cuanto más compleja y más reciente es una especie, más abundantes y más largos son los intrones de su genoma. Parece, por tanto, que los intrones constituyen una ventaja evolutiva, ya que estos fragmentos favorecen la recombinación meiótica al aumentar la distancia entre los exones.
Se distinguen tres tipos de ADN: ADN altamente repetitivo (ADN satélite). Constituye el 10% del total,
aunque puede llegar al 50%. Está constituido por secuencias cortas de 5 y 10 pares de bases que se disponen en tándem (una detrás de otra), y que llegan a
4
4

repetirse de 106 a 108 veces. Se sitúa en los extremos (telómeros) y en las constricciones (centrómeros) de los cromosomas, que son zonas genéticamente inactivas, es decir, que no se transcriben nunca. Puede que su única función sea mecánica, como el emparejamiento y la segregación de los cromosomas durante la mitosis y la meiosis.
ADN moderadamente repetitivo. Constituye el 20% del total. Está constituido por diversas secuencias de 150 a 300 nucleótidos que pueden disponerse en tándem o dispersas por todo el ADN. El número de copias varía entre 10 a más de 1.000 veces, pudiendo llegar en algún caso a más de 100.000. Comprende genes que codifican histonas, ARNr, ARNt y secuencias de función desconocida.
ADN no repetitivo o simple. Constituye el 70% del total. Contiene la mayor parte de la información que pasa al ARNm y da lugar a proteínas, y al ADN que está intercalado y que no se transcribe (ADN espaciador).
Una parte del genoma eucariota se encuentra en los cloroplastos y en las mitocondrias, se trata de moléculas circulares de ADN relativamente pequeñas, que carecen de histonas. Su estructura es parecida a la del cromosoma bacteriano. Los cromosomas de estos orgánulos forman un sistema genético propio con plena capacidad para realizar la replicación del ADN, transcripción y síntesis proteica.
El proyecto genoma humanoA finales de los años 80 se propuso el objetivo internacional de conocer la
secuencia de nucleótidos (3 x 109 pares de nucleótidos) de los genes del ser humano que están contenidos en los 23 pares de cromosomas. En 1990 se inició el Proyecto Genoma Humano.
Los objetivos del proyecto son: Elaborar un “mapa genético” para determinar en qué cromosomas y en qué
lugar de los mismos se encuentran cada uno de los genes humanos. Determinar la secuencia exacta de nucleótidos de cada gen para conocer la
proteína que codifica y sus posibles alteraciones. 3 billones de bases nitrogenadas. Se realiza fundamentalmente mediante la electroforesis en geles de distintos fragmentos de ADN y la ayuda de ordenadores.
Acumular la información en bases de datos. Desarrollar de modo rápido y eficiente tecnologías de secuenciación. Desarrollar herramientas para análisis de datos. Dirigir las cuestiones éticas, legales y sociales que se deriven del proyecto.Se ha conseguido secuenciar el ADN en un 99%, del cual un 85% se ha establecido
de manera precisa (12 del 2 de 2001), en el 2003 se ha producido el mapeo casi completo del mismo. Obtener la secuencia de un ser humano no sirve aún para conocer cuál es la misión que desempeñan los genes, simplemente identifica los “nucleótidos” que conforman la espiral del ADN.Pero interpretar el genoma llevará tiempo. Ahora se ha logrado la secuencia, falta por saber qué función tiene cada gen y cómo se interrelacionan entre ellos y cuál es su relación y en qué medida con las enfermedades.El genoma humano: aproximadamente 30.000 genes. Esta cifra de genes es sólo dos o tres veces mayor que el encontrado en el genoma de Drosophila, la mosca de la fruta y que tenemos genes comunes con bacterias y que no han sido encontrados en nuestros ancestros.
De un 40% de los genes identificados se desconoce su función. Menos del 5% de la secuencia contiene genes portadores de instrucciones para hacer proteínas. Más del 90% de la secuencia es exacta en un 99,99% (entre una persona y otra el ADN difiere sólo en un 0,2%). Los genes humanos son pocos y se encuentran alejados entre sí. Además, se encuentran muy fragmentados. La fragmentación de los genes humanos hace posible que se construyan muchas proteínas distintas a partir de los mismos genes, mediante la combinación de las instrucciones de formas diversas. Según parece, como mínimo el 35% de todos los genes humanos pueden leerse de muchas
5
5

formas. De esta manera, el genoma humano podría codificar cinco veces más proteínas que los genomas menos flexibles de la mosca del vinagre o del gusano.
Grandes tramos del genoma humano parecen haber sido antiguamente virus, puede que hace millones de años. Hay cientos de otros genes, que codifican un mínimo de 223 proteínas, que pueden proceder de las bacterias.
Analizado el genoma de unos pocos centenares de personas, los científicos han creado un catálogo bastante completo de la variación genética entre unos individuos y otros. En estas variaciones radica la clave de las diferencias hereditarias entre personas, incluyendo sus distintas propensiones a una u otra enfermedad.
Casi toda la variabilidad genética humana se basa en las mínimas alteraciones concebibles: las diferencias en una sola letra (nucleótido) en el ADN. Estas alteraciones en un solo nucleótido se denominan snips. Se han detectado 1,42 millones de snips. Las diferencias entre una persona y otra se deben a la combinación particular de snips tomados de ese conjunto básico de 1,4 millones.
Como el 95% del genoma es basura, la gran mayoría de esos snips están en el vasto desierto del ADN sin sentido. Pero, aún así, hay 60.000 snips que caen dentro de los genes.Aplicaciones futuras: La información contenida en los genes ha sido descodificada y permite a la ciencia conocer mediante test genéticos, qué enfermedades podrá sufrir una persona en su vida. El ADN encierra las claves para combatir una gran parte de las enfermedades, como el cáncer, la diabetes, la obesidad, los trastornos del sistema inmunológico y las degeneraciones nerviosas y cerebrales. Se podrán tratar enfermedades hasta ahora incurables.Diagnóstico precoz: Permitirán predecir qué personas son propensas a padecer determinadas enfermedades, por lo que se podrá avanzar en su diagnóstico y en su posterior tratamiento.Cáncer: La formación de un tumor se debe a una compleja acumulación de mutaciones en una sola célula de una persona, que provocan que la célula escape de control y prolifere indebidamente. Algunas de estas mutaciones ocurren durante la vida del individuo, causadas por agresiones externas como el humo del tabaco o la radiación ultravioleta de la luz solar. Otras mutaciones las lleva puestas cada individuo de nacimiento, lo que explica que algunas personas sean más susceptibles de desarrollar uno u otro tipo de cáncer. Conocer la combinación exacta de mutaciones en un tumor concreto de un paciente concreto (una combinación que determina estrictamente el comportamiento del tumor y permite predecir si va a responder aun fármaco o a otro) será pronto la herramienta básica para predecir el tratamiento óptimo de ese tumor.Enfermedades simples: De los 30.000 genes humanos, hay 1.100 cuyas variaciones (snips) se asocian a unas 1.500 enfermedades (algunos genes pueden mutar de varias formas para dar lugar a enfermedades clínicamente distintas).Enfermedades complejas: La propensión genética a las enfermedades más comunes, sin embargo, no se debe a la alteración de un solo gen, sino a decenas o cientos de genes simultáneamente.Fármacos a medida: Cada persona responde de forma muy distinta a cada fármaco. El análisis del genoma de cada persona permitirá predecir a qué fármacos va a sufrir reacciones peligrosas, o a cuál va a responder de manera óptima. El estudio del genoma, permitirá desarrollar medicamentos que modifiquen las funciones incorrectas de las proteínas, la verdadera causa de las enfermedades. También será posible elaborar medicamentos a la carta, específicos para cada persona.Envejecimiento: la manipulación de los genes responsables del envejecimiento aumentará la esperanza media de vida en cien años o más.Mapas individuales: En un plazo aproximado de veinte años, cada persona podrá tener su mapa genético individualizado, de tal modo que sabrá cuáles son sus puntos débiles constitucionales y su propensión a padecer enfermedades.
6
6

Se podrá informar a una persona, que puede comer alimentos grasos porque carece de predisposición genética a la obesidad y a enfermedades cardíacas, pero que debe huir del alcohol porque es genéticamente propenso al alcoholismo.En identificación forense, para identificar victimas de catástrofes, paternidad y otras relaciones familiares, identificar y proteger especies en peligro, detectar bacterias que pueden polucionar agua, aire, alimentos, determinar compatibilidad de órganos donantes en programas de transplante.En agricultura, ganadería y bioprocesamientos, se utiliza para mejorar la resistencia de cultivos ante insectos, sequías, para hacerlos más productivos y saludables, para producir animales más saludables y nutritivos, elaborar biopesticidas, vacunas comestibles y nueva limpieza del medio ambiente de plantas como tabaco.
Pero el conocimiento del código de un genoma abre las puertas para nuevos conflictos ético-morales, por ejemplo, seleccionar que bebes van a nacer, o clonar seres por su perfección. Esto atentaría contra la diversidad biológica. Quienes tengan desventaja genética quedarían excluidos de los trabajos, compañías de seguro, seguro social, etc.
Otro problema ético que se ha generado a partir del PGH, es el patentamiento de genes y secuencias génicas por parte de compañías biotecnológicas, universidades y gobiernos.
La duplicación del ADNLa necesidad de la duplicación del ADN
El ADN portador de la información genética debe transmitirse fielmente a cada una de las células hijas obtenidas tras la división celular. Por tanto, antes de producirse esta, es imprescindible que el ADN puede formar réplicas exactas de si mismo para disponer de dos copias iguales. Este proceso conocido como replicación o autoduplicación, resulta fundamental para asegurar que todas las células de un organismo pluricelular mantienen la misma identidad.
El mecanismo general de la replicación fue intuido por Watson y Crick cuando establecieron la estructura de doble hélice y la complementariedad de las bases. Propusieron que la doble hélice del ADN se abre y las dos cadenas de nucleótidos se separan; a partir de cada una de las dos cadenas se forma una nueva, que es complementaria de la que le ha servido como patrón.
La replicación o duplicación del ADN consiste en sintetizar, a partir de una molécula inicial (molécula madre), dos idénticas entre sí (moléculas hijas). La replicación permite a las células hijas contener el mismo ADN que la célula madre. Así, la información genética se transfiere desde una célula progenitora a toda su descendencia.
Primeras hipótesis sobre la duplicación del ADNLa estructura del ADN en doble hélice permite comprender cómo dicha molécula
es idónea para dar lugar a copias. Por un lado su estructura presenta dos cadenas complementarias entrelazadas, lo que se da gran estabilidad, y por otro, bastaría con que una enzima específica las separara para que cada una de ellas pudiera servir como molde para sintetizar, a partir de nucleótidos sueltos y bajo la acción de otra enzima, la hebra complementaria.
Se propusieron tres hipótesis: la hipótesis semiconservativa, la hipótesis conservativa y la hipótesis dispersiva.
La hipótesis semiconservativa sobre la duplicación del ADN se debe a Watson y Crick. En ella se sostiene que en las dos nuevas moléculas de ADN de doble hélice producidas, una de las hebras sería la antigua, que actuaría como molde, y la otra la
7
7

moderna, constituida por la polimerización de nucleótidos libres sobre ese molde, por complementariedad de las bases nitrogenadas.
En la hipótesis conservativa se propone que tras la duplicación quedan, por un lado, las dos hebras antiguas juntas y, por otro, las dos hebras nuevas también espiralizadas.
En la hipótesis dispersiva se propone que las hebras, al final, están constituidas por fragmentos distintos de ADN antiguo y de ADN recién sintetizado.
El experimento más definitivo para dilucidar cuál de estas tres hipótesis era la correcta fue el de Meselson y Stahl en 1957. La hipótesis confirmada fue la semiconservativa.Experimento de Meselson y Stahl
Meselson y Stahl cultivaron bacterias (Escherichia coli) en un medio con nitrógeno pesado (N15). El N15 es un isótopo del nitrógeno normal o N14. Tiene el mismo número de electrones (7 e) y de protones (7 p) que el átomo de N14, y así tiene el mismo comportamiento químico, pero posee un neutrón más (8 n), por lo que pesa más que el N14. Esto conlleva a que las moléculas de ADN sintetizadas con N15 pesen más que las construidas con N14, lo que permite separarlas mediante centrifugación utilizando un medio que presente gradiente de densidades. Las moléculas de ADN ligero quedan más arriba y las de ADN pesado más abajo en el tubo de centrífuga. Para realizar el experimento pasaron las bacterias cultivadas con N15 a un medio con nitrógeno normal (N14) durante una media hora, que es tiempo necesario para que se duplique el ADN bacteriano, lo extrajeron y lo centrifugaron. Luego, mediante rayos ultravioleta, que son muy absorbidos cuando atraviesan una disolución que contiene ADN, se pudo deducir que el ADN recién sintetizado ocupaba, en el tubo de centrifugación, una posición que era intermedia entre la que ocupaba el ADN con N15 y el ADN con N14. Se trataba, pues, de un AD híbrido y había que descartar la hipótesis conservativa.
Si en lugar de dejar las bacterias en N14 durante una división las dejaban durante dos, aparecían dos ADN, uno híbrido y otro ligero. Si se dejaban durante tres, el ADN híbrido era, en proporciones, mucho menos importante. Esto descartaba la hipótesis dispersiva y demostraba la hipótesis semiconservativa.
Meselson y Stahl completaron el experimento con otro ensayo. Después de aislar el ADN híbrido y someterlo a una temperatura de 80 a 100ºC durante 30 minutos, consiguieron separar las dos hebras y, mediante enfriamiento súbito, el que no se volvieran a asociar de nuevo. Posteriormente, mediante centrifugación del preparado,
8
8

comprobaron que una hebra era ligera y la otra densa, lo que confirmó definitivamente la hipótesis semiconservativa.
La síntesis del ADN in vitroEn 1956 Kornberg aisló a partir de la bacteria Escherichia coli la enzima ADN-
polimerasa. Esta enzima es capaz de sintetizar ADN in vitro.Para su actuación, la ADN-polimerasa necesita la presencia de
desoxirribonucleicos-5-trifosfatos de adenina, timina, guanina y citosina, iones magnesio Mg2+, y un ADN en el que una de las hebras actúa como hebra “patrón”, y un extremo de la otra, que se ha retirado en parte, como “cebador”.
La ADN-polimerasa es incapaz de iniciar una cadena de novo. Su papel se limita a añadir nucleótidos al extremo de una cadena preexistente, el llamado ADN cebador. Concretamente, actúa añadiendo nucleótidos al extremo que presenta libre el carbono 3´ del nucleótido, siendo incapaz de añadirlos en el otro extremo, que es el que presenta libre el carbono 5´ del nucleótido.
Así pues, la cadena del ADN cebador sólo puede crecer en el sentido 5´ 3´. Por ello, en una cadena de ADN, el nucleótido que tiene su carbono 5´ libre es el primer nucleótido de la cadena y el que tiene libre el carbono 3´ es el último nucleótido añadido y al que se puede añadir otro.
La ADN-polimerasa, además, actúa de forma que la nueva hebra sintetizada es antiparalela y complementaria.
Duplicación del ADN in vivoEntre los primeros experimentos encaminados a observar la duplicación del ADN
en los seres vivos cabe destacar el realizado por Cairns en 1963. El experimento de Cairns volvió a confirmar la hipótesis semiconservativa y, además, descubrió la existencia de un punto concreto como origen de replicación del ADN bacteriano. La replicación es bidireccional, es decir, hay una horquilla a la izquierda del punto de inicio y otra horquilla a la derecha, que van progresando en direcciones opuestas.
Se plantearon dos dilemas:El primero, era cómo la ADN-
polimerasa podía sintetizar sin necesidad de cebador; y el segundo, cómo las dos nuevas hebras de la horquilla crecían en paralelo: si una de ellas lo estaba haciendo en dirección 5´ 3´, la otra lo debía hacer en dirección 3´ 5´, lo cual era imposible de explicar, ya que ninguna ADN-polimerasa trabaja en esa dirección. La ADN-polimerasa recorre las hebras molde en sentido 3´ 5´ y va uniendo los nuevos nucleótidos en el extremo 3´ ( dirección 5´ 3´). Sin embargo, como las dos cadenas del ADN son antiparalelas, cuando se forma la horquilla de replicación la ADN-polimerasa solo puede sintetizar nucleótidos en uno de los dos sentidos. La síntesis de la nueva hebra orientada en sentido 3´ 5´ se realiza sin interrupción, a esta hebra se la denomina conductora o lider. El mecanismo de
9
9

síntesis de la hebra orientada en sentido 5´ 3´ (hebra retardada) fue descubierto por Okazaki.
La solución al dilema la dio el descubrimiento en 1968, por Okazaki, de unos fragmentos constituidos por unos 50 nucleótidos de ARN y unos 1.000 o 2.000 nucleótidos de ADN. Estos fragmentos, denominados, fragmentos de Okazaki, son sintetizados por la ARN-polimerasa, que no precisa cebador para empezar, y luego por la ADN-polimerasa, en dirección 5´ 3´ sobre diferentes regiones de la hebra patrón. Luego, sin moverse, tras perder su porción de ARN, pueden ser fusionados entre sí, pudiendo dar la sensación de que la nueva hebra de ADN crece en dirección 3´ 5´.
La hebra retardada se sintetiza de forma discontinua, es decir, se sintetizan pequeños fragmentos de forma separada y después se unen.
La replicación es semidiscontinua: en una cadena (llamada conductora) se sintetizan fragmentos bastante grandes de forma continua, mientras que en la otra (llamada retardada) la síntesis es discontinua, es decir, se sintetizan pequeños fragmentos de forma separada y después se unen.En bacterias:
Iniciación: desenrrollamiento y apertura de la doble hélice1. Existe una secuencia de nucleótidos en el ADN llamada “origen de la
replicación” (donde abundan las secuencias de bases GATC), que actúa como señal de iniciación. El punto de iniciación es reconocido por una proteínas que se unen a el.
2. El proceso se inicia con una enzima denominada helicasa que rompe los puentes de hidrógeno entre las dos hebras complementarias y las separa para que sirvan de patrones o moldes. Como el desenrollamiento de la doble hélice da lugar a superenrollamientos en el resto de la molécula, capaces de detener el proceso, se hace preciso el concurso de topoisomerasas que eliminen las tensiones en la fibra. Estas proteínas actúan cortando una (las topoisomerasas I) o las dos fibras (las topoisomerasas II) y, una vez eliminadas las tensiones, empalmándolas nuevamente. La topoisomerasa II de Escherichia coli se denomina girasa.
3. A continuación intervienen unas proteínas que se enlazan sobre el ADN de hebra única. Son las proteínas estabilizadoras (SSB), que tienen como función mantener la separación de las dos hebras complementarias. Se inicia así la formación de la horquilla de replicación.
4. El proceso es bidireccional, es decir, hay una helicasa trabajando en un sentido y otra trabajando en sentido opuesto. Las dos horquillas de replicación forman las llamadas burbujas u ojos de replicación.
Elongación: se sintetiza la nueva hebra de ADN sobre cada hebra de la doble hélice original.
10
10

Además de las enzimas que actúan en la iniciación, en la elongación intervienen las ADNpol I, II, III. Durante la elongación se lleva a cabo la corrección de errores que se hayan podido producir.
5. Como ninguna ADN-polimerasa puede actuar sin cebador, interviene primero una ARN-polimerasa que sí lo puede hacer. Esta encima se denomina primasa y sintetiza un corto fragmento de ARN de unos diez nucleótidos, denominado primer, que actúa como cebador, con un extremo hidroxílo 3´libre al que añadir los nuevos nucleótidos; una vez iniciada la síntesis, la propia cadena de ADN ya sintetizada actúa como cebador.
6. Interviene después la ADN-polimerasa III, que, partiendo del primer, comienza a sintetizar en dirección 5´ 3´, como todas las polimerasas, una hebra de ADN a partir de nucleótidos trifosfato. La energía necesaria para el proceso es aportada por los propios nucleótidos, que pierden dos de sus grupos fosfato. Esta nueva hebra es de crecimiento continuo, ya que la helicasa no se detiene, y se denomina hebra conductora.
7. S o b r e l a o t r a h e b r a q u e S o b r e l a o t r a h e b r a q u e e s a n t i p a r a l e l a , l a A R N -
polimerasa sintetiza unos 40 nucleótidos de ARN en un punto que dista unos 1.000 nucleótidos de la señal de iniciación. A partir de ellos, la ADN-polimerasa III sintetiza unos 1.000 nucleótidos de ADN, formándose entonces un fragmento de Okazaki. Este proceso se va repitiendo a medida que se van separando las dos hebras patrón. Posteriormente, interviene la ADN-polimerasa I (ADN-polimerasa aislada por Kornberg), que, primero, gracias a su función exonucleasa, retira los segmentos de ARN, y luego, gracias a su función polimerasa, rellena los huecos con nucleótidos de ADN. Aunque el mecanismo de corrección es muy eficaz, a veces queda alguno sin corregir. Finalmente interviene la ADN-ligasa, que empalma entre sí los diferentes fragmentos (cataliza la formación del enlace fosfodiester entre un extrema 3´OH y uno 5´fosfato de un ADN situados adyacentes uno del otro). Esta hebra es, pues, de crecimiento discontinuo. Como precisa que se desespiralice un segmento de varios miles de nucleótidos para que se inicie su síntesis, tarda más en crecer que la otra, y , por ello, se la denomina hebra retardada.
Durante la replicación es frecuente que se produzcan errores y se incorporen nucleótidos que no tengan correctamente apareadas sus bases. La
11
11

ADNpol I actúa entonces como exonucleasa y elimina los nucleótidos mal apareados.
Aunque el mecanismo de corrección de errores es muy eficiente, a veces queda alguno sin corregir. Esos errores pueden ser importantes en la evolución.
8. El proceso continúa así hasta la duplicación total del ADN.En eucariotas:
El proceso es similar al que se sigue en las bacterias. Cabe destacar dos diferencias básicas. La primera gran diferencia es que el ADN de los eucariontes está
fuertemente asociado a histonas, formando los nucleosomas. Esto dificulta la procesividad de las ADN polimerasas y, además, requiere que la replicación esté coordinada con la síntesis de histonas. Se ha observado que, durante la replicación, la hebra que sirve de patrón a la hebra conductora se queda con las histonas y ambas se enrollan sobre los octámeros antiguos. La hebra que sirve de patrón a la retardada y la retardada se arrollan sobre nuevos octámeros de histonas que llegan a los lugares de replicación para formar nuevos nucleosomas.
La segunda gran diferencia es que, teniendo en cuenta que la longitud del ADN de un cromosoma eucariótico es mucho mayor que el ADN bacteriano y que, seguramente debido a la presencia de histonas, el proceso es bastante más lento (unas diez veces más lento), en cada ADN de un cromosoma no hay un solo origen de replicación, sino aproximadamente un centenar. Se forman unas cien burbujas de replicación. Su distribución es irregular, pudiendo haber regiones con muchas burbujas y regiones con pocas. Se activan de forma coordinada y constituyen las llamadas unidades de replicación o replicones.
También cabe señalar que los fragmentos de Okazaki son más pequeños, de unos cien a doscientos nucleótidos (en procariotas de unos 1000 a 2000 nucleótidos).
La replicación en los procariotas tiene lugar antes de la división celular en el citosol, y en eucariotas el proceso de replicación se realiza durante el período S de la interfase, que dura unas seis a ocho horas.
El proceso de replicación del ADN se va completando normalmente hasta llegar al extremo del cromosoma, el telómero. Cuando se elimina el último ARNcebador la hebra retardada quedará incompleta, ya que la ADNpol no podrá rellenar el hueco, al ser incapaz de sintetizar en dirección 3´ 5´. Este hecho hace que el telómero se vaya acortando un poco cada vez que la célula se divide, fenómeno que se asocia a los procesos de envejecimiento y muerte celular. Al cabo de un número determinado de divisiones, se producirá una pérdida de una cantidad importante de material genético que provocará la muerte celular.
Las cadenas de ADN de los cromosomas eucariotas son lineales, no anulares, lo que plantea un problema añadido. Cuando se elimina el ARN cebador del extremo 5´ de cada una de las hebras recién sintetizadas, el hueco que queda no lo pueden rellenar las enzimas ADNpol III porque no encuentran extremos hidroxilos 3´ libres sobre los que adicionar nuevos nucleótidos. Esta imposibilidad de replicar los extremos de cada cromosoma hace que el telómero se vaya acortando en cada etapa de replicación con el envejecimiento y la muerte de las células: al cabo de un número
12
12

determinado de divisiones, tarde o temprano, se producirá la pérdida de una cantidad importante de material genético que provocará la muerte celular.
El ADN telomérico está constituido por secuencias de 6 nucleótidos que se repiten numerosas veces. La hebra del extremo 3´ es rica en guanina (AGGGTT en la especie humana) y, lógicamente las del extremo 5´ es rica en citosina. Se ha comprobado que, si los telómeros desaparecen, los cromosomas se vuelven inestables y se unen. La consecuencia de esta inestabilidad es la muerte celular.
Existe una enzima, llamada telomerasa (retrotranscriptasa o transcriptada inversa) y constituida por una parte proteica y un ribonucleótido (ARN), que actúa como molde para alargar la hebra del extremo 3´. Esta prolongación, catalizada por la telomerasa, sirve como cebador, por parte de la polimerasa, del extremo 5´ que quedó incompleto. La telomerasa aporta el molde, debido a que el telómero, está formado por una secuencia que se repite un gran número de veces. Así, la enzima solo necesita contener esta secuencia.
En las células que se dividen continuamente (células madres de los gametos, células embrionarias y células cancerígenas) hay una enzima llamada telomerasa que impide el acortamiento del telómero.
En las células somáticas la longitud del telómero disminuye a medida que se producen nuevas divisiones celulares. En los procariotas, sin embargo, el ADN es circular, y por lo tanto no hay telómeros, lo que significa que no se producen esos procesos de envejecimiento celular.
En los organismos unicelulares la actividad telomerasa evita el acortamiento de los telómeros. Los organismos unicelulares son inmortales, en el sentido de que no mueren inexorablemente, sino que se reproducen por divisiones sucesivas.
Los estudios realizados en células humanas han demostrado la existencia de la telomerasa en los tejidos germinales y embrionarios. No se ha detectado el enzima en muchos otros tejidos.
Los experimentos han demostrado la presencia de la actividad telomerasa en el 80% de los tumores malignos. Ni en las primeras etapas que dan lugar al tumor no en los tumores benignos se detecta telomerasa. Si se inhibiese la telomerasa, las células malignas perderían la posibilidad de dividirse indefinidamente, puesto que envejecerían.
Reacción en cadena de la polimerasaEs una técnica que permite duplicar un número ilimitado de veces un fragmento
de ADN en un tubo de ensayo. Mediante esta técnica pueden generarse millones de moléculas idénticas, a partir de una molécula de ADN. Esto se puede conseguir en unas horas.
Las enzimas ADN polimerasa duplican el ADN copiando las dos cadenas de la doble hélice. Debido a la acción de estas enzimas, a partir de una molécula de ADN bicatenario se obtienen dos moléculas de ADN bicatenario exactamente iguales.
Para que estas enzimas lleven a cabo su función correctamente debe existir:
13
13

Una cadena molde de ADN, que es una de las dos hebras de la doble hélice. Una mezcla de desoxiribonucleótidos trifosfato. Un cebador, que es un pequeño fragmento de ADN monocatenario, cuya
secuencia de bases es complementaria de uno de los extremos de la cadena molde.
Una fuente de calor.En 1985, Kary Mullis desarrolló la técnica llamada reacción en cadena de la
polimerasa, conocida también como PCR, que, a partir de la capacidad de la ADN polimerasa para replicar el ADN, permite obtener en el laboratorio múltiples copias de un fragmento determinado de ADN.
La reacción en cadena de la polimerasa se lleva a cabo de la siguiente forma. La molécula o fragmento de ADN que se desea replicar se desnaturaliza para separar las dos hebras de la doble hélice. Para ello se somete el ADN a temperaturas elevadas. Cada una de las dos hebras sirve como molde para sintetizar otra cadena complementaria. A fin de que la ADN polimerasa pueda actuar copiando cada una de las dos cadenas complementarias, son necesarios unos cebadores adecuados. En la mezcla de reacción debe existir, así mismo, una concentración suficiente de nucleótidos trifosfato, que componen las piezas de construcción de las cadenas que se van a sintetizar.
El proceso que se acaba de describir constituye un ciclo de síntesis. Si se repitiera el proceso íntegramente, se partiría de cuatro hebras molde y se obtendrían cuatro moléculas bicatenarias. Si se realiza un nuevo ciclo, se partiría de ocho hebras molde que, a su vez, darían ocho moléculas bicatenarias. En el siguiente ciclo, los moldes serían 16. Se trata, por tanto, de un proceso exponencial, en el que se llevan a cabo tantos ciclos como sea necesario para obtener el número de copias deseado. Tras 20 ciclos de síntesis se puede obtener del orden de un millón de copias de una molécula de ADN. Se utiliza la ADN polimerasa de una bacteria que vive en aguas termales (Thermus aquáticus), así la enzima puede trabajar a altas temperaturas.Aplicaciones de la PCR:
La finalidad de la PCR consiste en la amplificación del ADN, es decir, en la obtención de múltiples copias de un fragmento específico de ADN a partir de un número muy pequeño de moléculas.
Clonación de genes. La PCR ha resultado útil en la clonación de genes, ya que permite obtener un gran número de copias del gen antes de la clonación propiamente dicha. Gracias a esta técnica también es posible amplificar genes mutados artificialmente para su posterior clonación.
Estudios evolutivos. Mediante la PCR se pueden amplificar genes de organismos ya extinguidos a partir de cantidades muy pequeñas de ADN presentes en algunos fósiles, para compararlos posteriormente con genes semejantes de organismos actuales y estudiar la conservación de ciertos genes a lo largo de la evolución y reconstruir árboles filogenéticos.
Estudios históricos y arqueológicos. Se han podido conocer datos referentes a enfermedades de origen genético amplificando ADN de muestras de individuos momificados, pertenecientes a civilizaciones antiguas.
Huellas dactilares del ADN. Es posible comparar muestras diferentes de ADN para comprobar si pertenecen al mismo individuo o no, o si existe parentesco entre ellas. Esta técnica se aplica actualmente en medicina forense e investigaciones policiales, con el fin de identificar individuos a partir de muestras biológicas, como sangre, semen, piel o cabellos. También se utiliza en pruebas de paternidad.
Secuenciación: Mediante la PCR se pueden formar la suficiente cantidad de ADN molde para su secuenciación. Es mucho más sencillo y rápido que la clonación en células.
Transcripción
14
14

La transcripción es el paso de una secuencia de ADN a una secuencia de ARN, ya sea ARNm, ARNr o ARNt. Ocurre en el interior del núcleo. Para ello interviene una cadena de ADN que actúe como molde (de las dos cadenas de nucleótidos que forman el gen, solo una, la denominada molde, se transcribe realmente, mientras que la otra, llamada informativa, no lo hace), ribonucleótidos trifosfato de A, C, G y U, las ARN-polimerasas (ARNpol) y los cofactores () y rho ().
Las ARNpol recorren la cadena de ADN en sentido 3´ 5´ y añaden los nucleótidos complementarios a los de la cadena de ADN que se transcribe (cadena codificadora), la molécula de ARN crece en sentido 5´ 3´.
La transcripción es asimétrica: solamente se transcribe para cada gen una de las dos cadenas del ADN. La cadena transcrita se llama codificadora, y la cadena de ADN que no se transcribe se denomina estabilizadora.
En los organismos eucariotas, para cada gen, solamente se transcribe una cadena de ADN (la codificadora), de forma que genes distintos del mismo cromosoma pueden utilizar como codificadora una cadena diferente a la de otros genes.En bacterias
En bacterias existe solamente una ARN polimerasa que sintetiza todos los tipos de ARN.
En el caso de la síntesis de ARNm se distinguen las siguientes etapas:
1. Iniciación. En primer lugar, la ARN-polimerasa se asocia con el cofactor sigma (), que permite a esta enzima reconocer y asociarse a una región concreta del ADN denominada promotor, que a veces es común a varios genes. En esta región se han distinguido dos secuencias de consenso (secuencias cortas de bases nitrogenadas a las que se une la ARN-polimerasa), iguales o parecidas a TTGACA y a TATAAT, que se encuentran a distintas distancias antes del punto de inicio. Se han descubierto también secuencias intensificadoras, que favorecen la transcripción. A continuación, la ARN-polimerasa pasa de una configuración cerrada a una configuración abierta y desenrolla, aproximadamente, una vuelta de hélice permitiendo la polimerización de ARN a partir de sólo una de las hebras que se utiliza como patrón (la
15
15

ARN-polimerasa hace que la doble hélice de ADN se abra para permitir que quede expuesta la secuencia de bases del ADN y se puedan incorporar los ribonucleótidos que se van a unir). Posteriormente se separa el cofactor .
2. Elongación o alargamiento. El proceso continúa a razón de unos 40 nucleótidos por segundo. A medida que la ARN-polimerasa recorre la hebra de ADN patrón hacia su extremo 5´, se sintetiza una hebra de ARN en dirección 5´ 3´. Lee el ADN en sentido 3´ 5´, mientras que sintetiza en sentido 5´ 3´.
3. Finalización. La ARN-polimerasa reconoce en el ADN unas señales de terminación que indican el final de la transcripción. Esto implica el cierre de la burbuja formada por el ADN y la separación de la ARN-polimerasa del ARN transcrito. La finalización presenta dos variantes: una en la que se precisa el cofactor (que reconoce una secuencia específica del ARN y se une a ella para tirar del ARN y soltarlo de la ARN polimerasa), y otra en la que no se precisa: la finalización se produce al llegar a una secuencia palindrómica (secuencia que tiene la misma lectura de izquierda a derecha y de derecha a izquierda) rica en G y C, que posibilita la autocomplementariedad de la cola del ARN, lo que da lugar a un bucle final que provoca su separación del ADN. Entonces éste vuelve a formar la doble hélice y la ARN-polimerasa se separa.
4. Maduración. Si lo que se sintetiza es un ARNm, no hay maduración; cuando se transcribe ADN que codifica los ARNt y los ARNr se forma una larga molécula de ARN que contiene numerosas copias de las secuencias del ARNr o el ARNt. Esta larga molécula, el transcrito primario, es posteriormente cortada en fragmentos más pequeños por enzimas específicas, para dar lugar a los distintos ARNt y ARNr.
16
16

En eucariotas.Hay tres tipos de ARN-polimerasa, según el tipo de ARN que se ha de sintetizar.
ARN-pol I = ARNr ARN-pol II = ARNm ARN-pol III = ARNt
Los genes están fragmentados de forma que siempre es necesario un proceso de maduración en el que se eliminen las secuencias sin sentido o intrones y se empalmen las secuencias con sentido o exones. Excepcionalmente, hay genes, como los de las histonas, que no presentan intrones.
Se ha observado que en genes que se transcriben continuamente (como los de ARNr) el ADN está siempre extendido, en otros siempre está, aparentemente, en forma de nucleosomas, y en otros hay transición a la forma extendida sólo durante la transcripción.
En el caso de la síntesis de ARNm se distinguen las siguientes etapas:
1. Iniciación. Existe una región del ADN denominada región promotora, donde se fija la ARN-polimerasa II, que consta de dos señales denominadas secuencias de consenso: la CAAT y la TATA, a distintas distancias antes del punto de inicio.
2. Alargamiento o elongación. El proceso de síntesis continúa en sentido 5´ 3´. Al cabo de 30 nucleótidos transcritos se añade una caperuza constituida por una metil-guanosín-trifosfato invertida al extremo 5´.
3. Finalización. La finalización de la síntesis del ARNm parece ser que está relacionada con la secuencia TTATTT. A continuación interviene la enzima poli-A-polimerasa que añade al extremo final 3´ un segmento de unos 200 ribonucleótidos de adenina, la llamada cola de poli A, al transcrito primario o pre ARNm, también llamado ARN heterogéneo nuclear.
4. Maduración. En eucariotas la maduración es más compleja, ya que la mayor parte de los genes que codifican las proteínas están fragmentados. Cada gen
17
17

consta de varios fragmentos denominados intones (secuencias de bases más o menos largas que se transcriben, pero que no se traducen, es decir, no codifican una secuencia de aminoácidos) y exones (secuencias que se transcriben y se traducen), intercalados unos con otros. Se produce en el núcleo. La maduración consiste en la eliminación de los intrones y la unión de los exones. La maduración la realiza una enzima denominada ribonucleoproteína pequeña nuclear (RNPpn) que es un complejo proteína-ARNpn. El ARNpn contiene unas secuencias que son complementarias de las de los extremos de los intrones. Al asociarse, el intrón se curva y se desprende. A continuación, actúan ARN-ligasas específicas que empalman los exones. El ARNt y el ARNr presentan también procesos de maduración.
El código genéticoUna vez obtenida una copia del mensaje genético en forma de cadena de ARNm,
ésta dirige la síntesis de proteínas en los ribosomas. Para ello, estos orgánulos interpretan la secuencia concreta de nucleótidos existente en la molécula de ARNm
como la información necesaria para la unión de los aminoácidos precisos para construir la proteína específica.
Se denomina código genético a la relación entre la secuencia de nucleótidos (o, más concretamente, de bases nitrogenadas presentes en ellos) del ARNm y la secuencia de aminoácidos que constituye una proteína.
Consiste en una equivalencia entre dos polímeros específicos. Uno de ellos, el ARN, tiene dispuestas sus bases nitrogenadas en una secuencia concreta que contiene la información que determina el orden en que han de engancharse los sucesivos aminoácidos que forman la cadena polipeptídica. Por tanto, los ARNm con secuencias de bases nitrogenadas distintas llevan información para la síntesis de proteínas diferentes.
18
18

El código genético es la clave que permite la traducción del mensaje genético a su forma funcional, las proteínas. Como sólo hay cuatro bases nitrogenadas distintas, las señales codificadoras para los 20 aminoácidos proteicos deben estar constituidas por más de una base. Si la señal estuviera formada por dos bases nitrogenadas, sólo codificarían 42 = 16 aminoácidos, por lo que aún quedarían aminoácidos sin codificar. Por tanto, cada señal que codifica para un aminoácido está constituida por tres bases distintas (un triplete), es decir, 43 = 64 tripletes de bases distintas.
Los tripletes de bases del ARNm reciben el nombre de codones. Los tripletes del ADN correspondientes, que han sido transcritos, se denominan codógenos. Existen 61 codones codificadores de aminoácidos y 3 (UAA, UAG y UGA, llamados sin sentido) que señalan el final del mensaje y no especifican ningún aminoácido. Hay también un codón (AUG) que, además de codificar para el aminoácido metionina, es la señal de comienzo.
En la década de 1950 se emprendió la tarea de descifrar el código genético, es decir, de identificar la señal concreta en el ARNm para la introducción de cada aminoácido en la cadena proteica en formación. Severo Ochoa descubrió en 1955 la enzima polinucleótido fosforilasa, capaz de sintetizar ARN a partir de nucleótidos difosfato sin necesidad de un molde de ADN. Esto permitió sintetizar cadenas conocidas de ARN constituidas por un único tipo de nucleótido o por varios.
A raíz de sus trabajos con esa enzima en Escherichia coli, M. Nirenberg y H. Matthaei dedujeron en 1961 que el triplete de sus bases UUU codificaba para el aminoácido fenil-alanina, el CCC para la prolina y el AAA para lisina. Posteriormente se descifró el resto de los tripletes.
Colocaron una serie de 20 tubos con los veinte aminoácidos en cada uno de los tubos. En cada tubo había un aminoácido diferente marcado con C14. En todos y cada uno de los tubos añadió el poli U y todos los elementos necesarios para la síntesis proteica. Observó que solo aparecía un polipéptido radiactivo en el tubo donde había marcado la fenilalanina.
El código genético tiene las siguientes características: Está formado por una secuencia lineal de bases nitrogenadas. Carece de solapamiento. Los tripletes de bases se hallan dispuestos de
manera lineal y continua, sin que entre ellos existan espacios ni separaciones de ningún tipo, y sin que compartan ninguna base nitrogenada. Su lectura se hace en un solo sentido (5´ 3´), desde el codón que indica el comienzo de la proteína hasta el que indica su final. Sin embargo, existe la posibilidad de que un mismo ARNm contenga varios codones de iniciación. Esto significa que se podrían realizar varias fases de lectura y se sintetizaría más de un polipéptido.
19
19

Es universal. Es el mismo código para todas las células de todas las especies (incluso los seres acelulares como los virus emplean el mismo código). Sin embargo, se han detectado excepciones a la universalidad del código en el material genético de mitocondrias, en algunos protistas ciliados y en micoplasmas.
Es degenerado. El código está degenerado en el sentido matemático del término. Esto significa que no existe el mismo número de señales codificadoras en el ARN que aminoácidos van a ser codificados. Salvo el triptófano y la metionina, que están codificados por un único codón, los demás aminoácidos están codificados por más de un triplete. Generalmente, solo se diferencian en la última base. La existencia de codones distintos que codifican el mismo aminoácido, se puede considerar que proporciona cierta ventaja, puesto que si se produce un cambio no deseado en una base nitrogenada es posible que el codón alterado siga codificando para el mismo aminoácido.
No presenta imperfecciones. Ningún codón codifica más de un aminoácido.
TraducciónPara que tenga lugar el proceso de traducción o síntesis de proteínas se
necesitan: Ribosomas, donde se realiza la síntesis proteica. ARNm, que lleva la información para sintetizar cada proteína. Aminoácidos, que son los componentes de las proteínas. ARN de transferencia, que aporta los aminoácidos en el orden preciso. Enzimas y energía, necesarias en toda reacción de biosíntesis.La traducción se realiza en los ribosomas, orgánulos citoplasmáticos formados por
dos subunidades, una pequeña y otra grande, formadas por ARNr específicos y proteínas. En la subunidad pequeña se une el ARNm, mientras que en la subunidad grande es donde se unen los aminoácidos para formar la cadena polipeptídica. Ambas se unen cuando van a sintetizar proteínas.
En el ribosoma se distinguen tres lugares diferentes de unión a los ARN de transferencia: el sitio P (peptidil), donde se sitúa la cadena polipeptídica en formación; el sitio A (aminoacil), donde entran los aminoácidos que se van a unir a la cadena proteica, y el sitio E, donde se sitúa el ARNt antes de salir del ribosoma.
En la biosíntesis de las proteínas podemos distinguir las siguientes etapas:
1. Activación de los aminoácidos.2. Traducción: iniciación de la síntesis. Elongación o
alargamiento de la cadena polipeptídica. Finalización de la síntesis.
3. Asociación de varias cadenas polipeptídicas y, a veces, de grupos prostéticos, para constituir las proteínas.
20
20

1. Activación de los aminoácidos. Los aminoácidos, en presencia de la enzima aminoacil-ARNt-sintetasa y de ATP, son capaces de asociarse a un ARNt específico y dar lugar a un aminoacil-ARNt, liberándose AMP, Ppi y quedando libre la enzima, que vuelve a actuar. La unión del aminoácido a su ARNt específico se realiza entre su grupo carboxilo (-COOH) y el radical –OH del extremo 3´ del ARNt.
2. Primera etapa de la traducción: la iniciación de la síntesis. La traducción se inicia en el extremo 5´ del ARNm.
En
21
21

bacterias, el ARNm no experimenta maduración. Incluso antes de acabarse su síntesis ya se empieza a traducir. El ARNm se une a la subunidad pequeña de los ribosomas gracias a una secuencia inicial llamada región lider, que no se traduce, en la que hay unos 10 nucleótidos complementarios con el ARN ribosómico. Se une por su extremo 5´. Gracias a un factor proteico de iniciación IF3. (5´ AUG 3´). A éstos se asocia el aminoacil-ARNt, gracias a que el ARNt tiene, en una de sus asas, un triplete de nucleótidos denominado anticodón, que se asocia al primer triplete de codón del ARNm según la complementariedad de las bases (formación de puentes de hidrógeno entre las bases complementarias del anticodón-codón), posteriormente se une la subunidad grande, formándose el complejo ribosomal o complejo activo que posee dos sitios de unión o centros: el centro peptidil o centro P, donde se sitúa el primer aminoacil-ARNt, y el centro aceptor de nuevos aminoacil-ARNt o centro A. Todos los procesos están catalizados por los llamados factores de iniciación. Todos estos procesos precisan gasto de GTP. El primer triplete que se traduce es el AUG, que corresponde a la formilmetionina. Posteriormente este aminoácido suele ser retirado.
En células eucariotas, el ARNm es sintetizado en el núcleo y antes de salir experimenta un proceso de maduración. En el extremo 5´ lleva una caperuza constituida por una metil-guanosín-trifosfato, que permite su identificación por los ribosomas, y a continuación la llamada región líder, que no se traduce. Después está el triplete AUG que se traduce por el aminoácido metionina que, frecuentemente, después es retirado.
3. Segunda etapa de la traducción: elongación de la cadena polipeptídica. Al centro A llega el segundo aminoacil-ARNt. El radical carboxilo del aminoácido iniciador se une con el radical amino del aminoácido siguiente mediante enlace peptídico. Esta unión es catalizada por la enzima peptidil-transferasa. El centro P queda, pues, ocupado por un ARNt sin aminoácido que sale del ribosoma. Se produce entonces la translocación ribosomal (desplazamiento del ribosoma sobre el ARNm es en sentido 5´ 3´). Como este desplazamiento es exactamente de tres bases, el primer ARNt abandona el ribosoma, y el dipeptidil-ARNt, que todavía se mantiene unido a su codón, pasa a ocupar el centro P. Todo ello precisa de GTP y está catalizado por los factores de elongación. En estas condiciones, otro aminoacil -ARNt se puede incorporar al sitio A, de manera que el proceso de alargamiento de la cadena proteica puede continuar, repitiéndose el ciclo.
4. Tercera etapa de la traducción: la finalización de la síntesis. La terminación de la cadena proteica tiene lugar cuando el ribosoma llega a un lugar del ARNm donde se encuentra un codón de terminación (UAA, UGA o UAG), que no es reconocido por ningún ARNt (no existe ningún ARNt cuyo anticodón sea complementario de ellos) y si por unos factores proteicos de liberación (FR) que precisan gasto de GTP para actuar. Se instalan sobre el centro A y provocan que la peptidil-transferasa haga interaccionar el último grupo –COOH con el agua, con lo que queda liberada la cadena polipeptídica. A continuación, se separan el ARNm y las dos subunidades ribosomales.
22
22

Un mismo ARNm, si es lo suficientemente largo, puede ser traducido por varios ribosomas a la vez, uno detrás de otro formando un polirribosoma o polisoma.
5. Asociación de varias cadenas polipeptídicas y, a veces, de grupos prostéticos, para constituir las proteínas. A medida que se va sintetizando la cadena polipeptídica, ésta va adoptando una determinada estructura secundaria y terciaria. Tras finalizar la traducción hay proteínas enzimáticas que ya son activas y otras que precisan eliminar algunos aminoácidos para serlo. Generalmente, se produce la separación del aminoácido metionina o aminoácido iniciador. Algunas enzimas necesitan asociarse a iones o a coenzimas para ser activas.
Regulación de la expresión génicaLas células no están constantemente sintetizando todos los tipos de proteínas
sobre las cuales tienen información; depende de las necesidades celulares. Si fuera así, se produciría un caos metabólico y alteraciones importantes.
Para evitar el despilfarro de moléculas y energía, los genes sólo se expresan cuando es necesario sintetizar las proteínas adecuadas en cada momento de la vida celular.
Es evidente que debe existir un sistema de regulación. Como la cantidad de proteínas sintetizadas depende directamente del ARNm presente en el citoplasma y como la vida media de éste es muy corta, es, pues, suficiente con regular la síntesis del ARNm. Ésta depende, en los procariontes, del sustrato disponible, y en las células eucariotas de los organismos pluricelulares, del ambiente hormonal del medio interno.Regulación de la expresión génica en los procariontes
Uno de los modelos de regulación de la expresión génica mejor conocidos en procariontes es el modelo del operón, descrito en los años cincuenta por F. Jacob y J. Monod en Escherichia coli. De acuerdo con este modelo existen unas proteínas reguladoras que controlan la transcripción de los genes que codifican para las enzimas implicadas en una ruta metabólica determinada. Se distinguen cuatro tipos de genes:
23
23

Gen promotor. Es una secuencia de nucleótidos del ADN a la que se une la ARN-polimerasa para iniciar la transcripción de un gen o un conjunto de genes.
Genes estructurales. Son los que codifican (contienen la información) para la síntesis de las proteínas implicadas en un mismo proceso metabólico. Se transcriben sin interrupción, de modo que el ARNm resultante lleva la información para varias proteínas y recibe el nombre de ARNm policistrónico.
Gen operador. Secuencia de nucleótidos situada entre el promotor y los genes estructurales. Es el lugar del ADN donde puede unirse una proteína reguladora e impedir la transcripción de los genes estructurales.
Gen regulador. Sintetiza la proteína reguladora (represor). Puede estar situado en cualquier lugar del cromosoma bacteriano. Cuando la proteína represora se asocia al operador impide físicamente que la ARN-polimerasa se pueda unir al ADN y con ello imposibilita la transcripción. Cuando el represor se separa, la transcripción ya es posible.
Según se trate de una ruta catabólica o anabólica se diferencian dos tipos de sistemas de regulación de la expresión génica: inducible y represible.Sistema inducible:
24
24

Es característico de los procesos catabólicos. La proteína sintetizada por el gen regulador es un represor activo que, al unirse al gen operador, impide la acción de la ARN polimerasa sobre los genes estructurales. Por tanto, no se formarán las enzimas de la ruta metabólica y ésta no se podrá realizar. Cuando aparece el sustrato inicial de esta ruta, la proteína represora cambia su conformación volviéndose inactiva, y deja de actuar sobre el gen operador. La inactivación del represor se consigue por la unión a una molécula inductora, que puede ser el mismo sustrato o un derivado de éste, de modo que se favorece la acción de la ARN polimerasa sobre los genes estructurales, los cuales se transcriben y sintetizan las enzimas correspondientes.
Este mecanismo permite la síntesis de proteínas enzimáticas solamente cuando son necesarias. Hasta que no hay sustrato que catabolizar no existen las enzimas para hacerlo, pues el gen represor, unido al gen operador, impide la transcripción. Sin embargo, la presencia del sustrato inactiva al represor e induce la síntesis de las enzimas que llevan a cabo la ruta catabólica.
El operón lac (de lactosa) constituye un sistema inducible que permite la regulación de la síntesis de las enzimas que intervienen en el catabolismo de la lactosa por Escherichia coli. Existen tres genes estructurales que codifican para las tres enzimas que participan en la ruta catabólica. La alolactosa, derivada de la lactosa, es el inductor que modifica la estructura del represor inactivándolo.Sistema represible
Se da en procesos anabólicos. A diferencia del sistema inducible, el represor sintetizado por el gen regulador es inactivo y, por tanto, la ARN polimerasa actúa y los genes estructurales se transcriben. La molécula represora se activa únicamente al cambiar su conformación por la unión a un correpresor, el cual constituye el producto final de la ruta anabólica. La activación del represor permite su unión al gen operador impidiendo la transcripción de los genes estructurales. De esta forma se producen las enzimas que participan en la ruta anabólica hasta que existe suficiente cantidad de producto final. En este caso, el represor se hace activo y se inhibe la transcripción, con lo cual se evita una síntesis excesiva del producto.
Control de la biosíntesis proteica por el AMP cíclicoEl AMP cíclico se forma a partir del ATP por la enzima adenilato-ciclasa, que está
situada en la cara interna de la membrana citoplasmática: Adenilato ciclasaATP ------------------ AMPc + Ppi
El AMPc para actuar necesita el concurso de la proteína denominada proteína activador del catabolito (CAP). El complejo CAP-AMPc tiene una gran afinidad por una zona que hay en el promotor, anterior al lugar donde se sitúa la ARN-polimerasa. Parece ser que, sin su presencia, la ARN-polimerasa tiene grandes dificultades para asociarse a su lugar en la zona promotor.
Se ha comprobado que cuando aumenta el nivel de glucosa en la célula, disminuye el nivel de AMPc (por un mecanismo todavía desconocido). No se forma el
25
25

complejo CAP-AMPc, la ARN-polimerasa no se puede fijar, y, por tanto, no se producen las enzimas para el metabolismo de la lactosa. As pues, sólo cuando se agota la glucosa, y además hay presencia de lactosa, la bacteria produce las enzimas para metabolizar dicha lactosa.El control de la expresión génica en eucariontes
En los organismos pluricelulares las células de diferentes tejidos tienen distintas funciones y sintetizan distintas proteínas, aunque contengan los mismos genes. Aunque todas las células tienen el mismo ADN, no en todas se manifiesta la misma información. Por ejemplo, los genes de la hemoglobina solo se manifiestan en los eritrocitos, los genes de la melanina en las células epiteliales, etc. Los segmentos de ADN que se encuentran fuertemente condensados no se expresan, mientras que los que están extendidos, incluso sin formar nucleosomas y facilitando la acción de la ARN-polimerasa, serán los que se transcribirán. Según las zonas que quedan condensadas aparece, durante el desarrollo embrionario, la diferenciación celular y con ella la organogénesis o formación de órganos en el embrión. Según el tipo de célula, habrá unos receptores de membrana u otros y, por tanto, sólo unas serán células diana respecto a unas hormonas determinadas.
En el ADN de las células eucariotas, además de los genes estructurales que se transcriben, hay una serie de secuencias relacionadas con el control de la transcripción:
El promotor, al que se une la ARN polimerasa después de que se haya unido un factor de la transcripción (proteína activadora).
Las secuencias intensificadoras, donde se unen factores de la transcripción que la activan.
Las secuencias silenciadoras, donde se unen factores de la transcripción que la inhiben.Los factores de la transcripción son proteínas reguladoras que controlan
específicamente la expresión de cada gen, según la situación fisiológica de la célula.El funcionamiento de algunos factores de la transcripción está condicionado por
la presencia de hormonas, de diferente modo según sean hormonas lipídicas o proteicas.
Las hormonas lipídicas (esteroideas), debido a su bajo peso molecular y su liposolubilidad, atraviesan la membrana plasmática y se difunden en el citoplasma, donde se unen a sus receptores específicos formando complejos “hormona-receptor”, que las introducen en el núcleo. Allí se fijan sobre secuencias determinadas del ADN e inducen la transcripción de determinados genes, seguramente facilitando la descondensación de ciertas zonas de ADN.
Las hormonas proteicas no penetran en el medio interno de las células del órgano blanco, debido a su elevado peso molecular. Las hormonas proteicas al unirse a su receptor de membrana inducen la activación de una enzima, la adenilatociclasa asociada al receptor y situada en la cara interna de la membrana. La adenilatociclasa cataliza la transformación del ATP en AMP cíclico (AMPc) que actúa como segundo mensajero, el cual activa a su vez a una proteína reguladora de la transcripción.
26
26

Genética1.- ¿De qué tipo de moléculas está constituido el virus T2? ¿Por qué A. Hershey y M. Chase eligieron isótopos radiactivos de S y de P, y no de C, N, O o H, para realizar su experiencia?. ¿Qué función realiza la cápsida y cuál el ADN en el bacteriófago T2? ¿Por qué el experimento realizado por A. Hershey y M. Chase demuestra que las proteínas no aportan información genética a los descendientes?Observa la figura y comenta por qué una de las hipótesis se denomina conservativa y otra se denomina semiconservativa.En la figura , se observa la posición que ocupa un ADN sintetizado con N14, un ADN sintetizado con N15 y el ADN obtenido en la primera experiencia de Meselson y Stahl. Explica qué posición habría ocupado el nuevo ADN si la hipótesis conservativa fuera correcta y por qué se descartó dicha hipótesis.
2.- En la figura anterior tambien se observan los resultados tras la segunda y tercera replicación. Explica qué resultados se habrían obtenido si la hipótesis correcta fuera la dispersiva y por qué se rechazó dicha hipótesis.3.- ¿En qué consiste, básicamente, la duplicación o replicación del ADN y cuál es su importancia biológica? Describe el mecanismo de replicación del ADN, indicando qué quiere decir que es un proceso bidireccional.
27
27

4.-Explica brevemente cómo se produce el flujo de información genética en un organismo. Di en qué consiste cada uno de los procesos biológicos implicados en este flujo.5.- La ADN-polimerasa precisa nucleótidos trifosfato a pesar de que, una vez incorporados, quedan nucleótidos monofosfato. Sabiendo que los enlaces entre dos grupos fosfato son muy ricos en energía, ¿qué explicación puedes dar a este hecho?6.- La nueva hebra siempre crece en dirección 5’ 3’. ¿Qué quiere decir esto?7.- ¿Cuál es la secuencia de ADN complementario de: 5’ ... ACTCAGGTA ... 3’?8.- Si en un ADN de una célula eucariota hay un 18% de timina, ¿qué porcentaje hay de las otras bases nitrogenadas?. ¿Qué son los fragmentos de Okazaki?9.- Completa el siguiente cuadro, especificando la función de las siguientes enzimas, que intervienen en la duplicación del ADN bacteriano.
Enzima FunciónProcesos en los que interviene
ADN-polimerasa I
Topoisomerasa I y II
Primasa (ARN- pol)
ADN-ligasa
ADN-polimerasa III
Helicasa
10.- ¿Por qué la hebra retardada se sintetiza de forma discontinua? ¿Por qué la hebra retardada tarda más en sintetizarse si cuenta con más puntos de iniciación?11.- Después de extraer y desnaturalizar el ADN de bacterias (Escherichia coli) que estaban iniciando la replicación, Okazaki observó que había fragmentos de cadena sencilla y largos, de unos 1000 nucleótidos; si dejaba que la replicación progresara durante más tiempo, encontraba fragmentos largos de cadena sencilla y fragmentos cortos. ¿Qué relación pueden tener estos resultados con el hecho de que las ADN-polimerasas sólo actúan en sentido 5 ’ 3’?12.- Diferencia entre procariotas y eucariotas en la duplicación del ADN.13.- Sin consultar el libro, rotula las enzimas que intervienen en la duplicación del ADN bacteriano.
28
28

14.- Explica lo que significa la sentencia: “un gen, una enzima”.15.- Comenta la siguiente frase: El ARN m de los procariotas suele ser policistrónico mientras que el de los eucariotas es monocistrónico16.-Un fragmento de una cadena de ADN tiene la siguiente secuencia de nucleótidos:
5´... TCTGCATCCTT ... 3´a) Escribe la cadena complementaria tras la replicación del mismo.b) Supón que en la replicación anterior el punto de iniciación (origen de la replicación)
es el nucleótido adenina, que aparece subrayado en la cadena. - Desde dicho punto hacia la derecha indica si la síntesis es continua o discontinua. Razona la respuesta- Desde dicho punto hacia la izquierda indica si la síntesis es continua o discontinua. Razona la respuesta.
c) En el caso del fragmento cuya síntesis es discontinua indicar: cuál es el fragmento de Okazaki que primero se forma, si el más próximo al punto de iniciación o el más alejado. Cuál es la dirección de síntesis de cada fragmento de Okazaki.
17.- Escribe la secuencia de ARNm que se transcribiría de la siguiente cadena de ADN.ADN: 3’ ... TACAAGTACTTGTTTCTT ... 5’
18.- Escribe la secuencia de ARNm que se transcribiría de la siguiente cadena de ADN.ADN: 5’ ... TACAAGTACTTGTTTCTT ... 3’
19.- Realiza el dibujo de dos instantes sucesivos de una translocación ribosomal sobre un ARNm inventado. Consultar la clave genética.20.- Escribe la secuencia de ARNm que se transcribiría de la siguiente cadena de ADN bacteriano y la secuencia de aminoácidos que resultaría de su traducción.
5’ ... TACAAGTACTTGTTTCTT ...3’
Supón que las dos guaninas se cambian por citosinas. ¿Cómo afectarían estas mutaciones a la secuencia de aminoácidos?
Supón, ahora, que las dos guaninas se eliminan de la secuencia del ADN. ¿Cómo afectarían estas otras mutaciones a la secuencia de aminoácidos de la proteína?21.-
29
29

a) El esquema representa dos procesos biológicos muy importantes. ¿Cómo se
denominan? Indicar los distintos elementos de la figura representados con los números.
b) Identificar los extremos del elemento 2 (a,b) y los extremos del elemento 5 (c,d).c) A partir de los datos del esquema podría concluirse que estos procesos están
ocurriendo en una célula procariota. ¿Por qué?22.- Definir: codón, anticodón, gen, locus, alelo, telómero.23.- ¿Qué secuencias nucleotídicas de ADN pueden codificar el siguiente tripéptido?
H2N - lys – Met – Glu – COOH24.- Supongamos la hebra de ADN:
3’ ... AATACAAAT ...5’
Durante la transcripción de la misma hay un error de tal manera que frente al nucleótido de “C” se sitúa otro de citosina, en lugar de uno de guanina. ¿Se modificará la secuencia peptídica codificada por esa hebra de ADN?25.- ¿Cuál es la secuencia de un segmento de ADN de doble hélice que ha servido de molde para sintetizar el siguiente ARNm: 5’ ... AUCCUCAUG ... 3’?26.- Contestar SÍ o NO?.
Procariotas Eucariotas¿El ARN transcrito primario es ya el ARNm?
¿Se realiza la transcripción y la traducción en compartimentos distintos?¿La transcripción y la traducción son simultáneas?
27.- ¿Cómo se encuentra codificada la información genética? ¿Qué son los fragmentos de Okazaki, a que proceso biológico los asocias, y qué enzimas intervienen en su formación?28.-
En el esquema adjunto se representan los pasos para la introducción y expresión de un gen (gen A) de un organismo en otro. El producto del gen humano (proteína A) se expresa en el organismo receptor (bacteria) después de que le sea introducida la molécula transformada.
a) ¿Cómo se denomina el proceso por el que se forman las proteínas codificadas por el gen A?
b) ¿Qué molécula se formaría a partir del gen A antes de que su información se convierta en proteínas? ¿Cómo se denomina este proceso? Sin entrar en detalles moleculares, descríbelo.
30
30

c) ¿Qué características tiene el código genético que permite expresar un gen de origen humano en un organismo tan distinto como una bacteria? ¿Qué otras características tiene el código genético?
d) ¿Cuál fue la aportación de Severo Ochoa al descubrimiento del código genético?29.- ¿Qué diferencia ves entre inducción y represión? ¿Qué semejanza existe entre ambos procesos de regulación génica?30.- ¿Qué diferencia hay entre operón y operador?31.- Indica la relación existente entre los distintos tipos de genes y su función:
1) Gen regulador. a) A él se une la ARN-polimerasa.2) Gen promotor. b) Codifica la síntesis de proteínas enzimáticas o no.3) Gen operador. c) Codifica la síntesis del represor.4) Gen estructural. d) A él se une el represor o complejo activo.
32.- El operón está formado por:a) Una secuencia de genes reguladores unidas al gen promotor.b) Una secuencia de genes operadores unidos al gen regulador.c) Una secuencia de genes estructurales unidos a un gen promotor y un gen operador.d) Una secuencia de genes estructurales unidos al represor activo y al gen operador.
33.- Indica cual de estas características corresponde a una inducción enzimática según la teoría del operón:a) La presencia de un compuesto hace que éste se una al represor activo y forme un complejo inactivo que no se une al gen operador.b) La presencia de un compuesto hace que éste se una al represor inactivo y forme un complejo activo que se une al gen operador.c) La presencia de un compuesto hace que éste actúe sobre el ATP formando el AMP-cíclico que actúa sobre el represor.
34.- Además de la inducción y represión, en los eucariotas pueden existir estos dos tipos de regulación génica:a) …………………………………………..…
b) …………………………………………..…35.- ¿Qué hormonas son las que actúan formando un segundo mensajero y cuál es éste?36.- Explica las razones por las cuales una mutación puntual en un determinado gen (sustitución de un nucleótido de la secuencia del ADN por otro) puede conducir a la producción de una enzima no funcional como producto de la traducción de dicho gen. ¿Por qué razón muchas mutaciones puntuales tienen efecto nulo?37.- Indica la relación existente entre los tipos de mutación y su causa:
1) Mutación cromosómica. a) Cambios en la secuencia de nucleótidos de un gen.2) Mutación génica. b) Cambio en la estructura de uno o varios cromosomas.3) Mutación genómica. c) Cambio en el número de cromosomas.
38.- Las mutaciones genómicas pueden ser:a) Euploidías d) Aneuploidíasb) Delecciones e) Inversionesc) Puntuales
39.- Indica la relación existente entre el tipo de mutación y su definición:1) Duplicación. a) Cambio de sentido de un segmento cromosómico dentro del
cromosoma.2) Transposición. b) Pérdida de parte de un cromosoma.3) Delección. c) Paso de un segmento de un cromosoma a otro cromosoma.4) Inversión. d) Repetición de un segmento cromosómico.
40.- Indica qué conceptos relativos a las mutaciones están relacionados:1) Aneuploidía. a) Transversión.2) Puntual. b) Paracéntrica.3) Delección. c) Poliploidía.4) Euploidía. d) Transposición.5) Translocación. e) Trisomía.6) Inversión. f) Deficiencia.7) Puntual. g) Sustitución.
41.- El origen y la evolución de las especies biológicas se deben a ……………………
31
31

42.- ¿Conoces algún otro tipo de regulación génica en células eucariotas además del sistema inducción-represión? Razona la respuesta.43.- ¿Es lo mismo represor que correpresor? Razona la respuesta.44.- a) ¿Las mutaciones son alteraciones al azar o dirigidas hacia un cambio concreto?; b) ¿Las mutaciones, generalmente, mejoran o empeoran la información inicial?; c) ¿Por qué son la base de la evolución de las especies?; d) ¿Por qué en algunos casos la aparición de una mutación puntual puede ser causa de una enfermedad grave y otras veces no se expresa?, e) ¿Qué diferencia hay entre mutación a nivel molecular y a nivel cromosómico?45.- En una determinada hebra de ADN se han producido las siguientes alteraciones:
a) En lugar de una A hay una G,b) En lugar de una T hay una Cc) En vez de una G hay una Cd) Además, falta el nucleótido de C
¿Qué tipo de mutación es cada una de ellas? ¿Cuál es la más importante?46.- ¿En qué triplete de ARNm de los que codifican la arginina es más probable que la sustitución de una sola base codifique el triptófano?47.- ¿Qué crees que sucedería si en una secuencia de nucleótidos de un gen, se suprime una base? ¿Cómo quedarían los tripletes o codones? ¿Qué pasaría con la proteína codificada por ese gen? Supón que esta proteína es responsable de la visión de un organismo. ¿Es lo mismo que el cambio se dé en una célula cualquiera del cuerpo, o que se dé en los óvulos de ese organismo, suponiendo que fuese un animal hembra? ¿Qué ocurriría con la descendencia?48.- Indica qué diferencias existen entre un individuo trisómico y uno triploide. ¿Qué tipo de mutación aparece asociada al síndrome de Down?49.- ¿En qué consiste el proyecto Genoma Humano? ¿Qué ventajas nos aportará?50.- ¿Qué genes no codifican proteínas? ¿Qué son genes solapados?51.- ¿Cuál es la posible razón por la que los genes que codifican el ARNt, ARNr y las histonas se encuentran replicados hasta más de 100 veces, dispuestos unos detrás de otros?52.- ¿Cuál es la posible función de las secuencias cortas que se encuentran repetidas miles de veces, sobre todo en los centrómeros y en los telómeros?53.- ¿Cuál es la posible explicación evolutiva de que en las células eucariotas, el ADN que codifica proteínas no supere el 1% del total, es decir, que haya una enorme cantidad de ADN extra (ADN repetitivo, intrones, pseudogenes, etc)?54.- ¿Qué ventajas tiene que no todas las réplicas de los genes duplicados sean iguales, sino que formen familias de genes algo diferentes?55.- ¿Qué tipo de vectores de clonación se utilizan para transferir genes a células procariotas y eucariotas? ¿Qué son los organismos transgénicos?56.- ¿Por qué en ingeniería genética es una ventaja utilizar como vector de genes un plásmido que posee información sobre resistencia a ciertos antibióticos?57.- ¿Por qué se utiliza la transcriptasa inversa vírica para introducir genes de células eucariotas en bacterias?58.- ¿Por qué es más fácil aplicar la ingeniería genética a los peces que a los mamíferos? ¿Qué es un organismo transgénico?59.- ¿Qué peligros respecto al equilibrio ecológico y respecto a la salud humana se pueden derivar de la ingeniería genética?60.- ¿Cómo se pueden obtener sustancias beneficiosas para el hombre (por ejemplo, insulina) mediante ingeniería genética?61.- ¿Conoces la función que desempeñan los enzimas de restricción? ¿Cómo se puede clonar un gen?62.- ¿Qué es la técnica de la PCR (reacción en cadena de la polimerasa)?. Indica alguna aplicación concreta de esta técnica y explícala brevemente.63.- Indica si son verdaderas o falsas:
1. El código genético es degenerado porque un mismo triplete codifica a varios aminoácidos.
2. La transcripción del ADN es el proceso mediante el cual el ADN forma copias exactas de si mismo.
3. La replicación del ADN es conservativa.
32
32

4. Los promotores son secuencias de ARN a las que se unen las ARNpol.5. Las encimas de restricción cortan el ADN.6. La traducción del ARNm tiene lugar en el núcleo.7. En los ribososmas se produce la transcripción.8. El intrón es la parte del gen eucariota que se transcribe y se traduce.9. Los tripletes de bases del ARNm se llaman codones.10.El proceso de la conjugación implica el paso de ADN de una bacteria a otra.11.Según el código genético, cada aminoácido se especifica por un codón que
consta de tres nucleótidos.12.Los plásmidos son moléculas de ADN circular de doble cadena.13.Las mutaciones son una fuente de variabilidad genética.14.Sólo una cadena de ADN actúa como molde en la síntesis del ARN durante la
transcripción.15.Locus es el lugar que ocupa un gen en el cromosoma.16.La síntesis de la cadena polipeptídica finaliza cuando el ribosoma reconoce el
codon teminador17.Los plásmidos son vectores que se emplean en ingeniería genética.18.La reacción de polimerización en cadena (PCR) permite la amplificación de
moléculas de ARN.19.En las células eucariotas y procariotas el ADN tiene siempre estructura
bicatenaria.20.El sentido de crecimiento de la cadena de ADN durante la replicación es siempre
en sentido 3´ a 5´.21.El proceso de transcripción del ARNm se realiza en el citoplasma.22.Una cadena de ARN mensajero puede ser leída simultáneamente por varios
ribosomas.23.En el proceso de la traducción la información genética de una cadena de ARNm
se transforma en una cadena polipeptídica.24.En la maduración del ARNm se eliminan los trozos correspondientes a los
intrones.25.El genotipo es consecuencia de la interacción entre el fenotipo y el ambiente.26.La insulina se puede obtener a partir de bacterias modificadas genéticamente. 27.La maduración del ARNm tiene lugar en el núcleo.28.En los ribosomas se produce la transcripción.
64.- Relaciona los siguientes científicos con sus descubrimientos: Darwin, Pasteur, Krebs, Severo Ochoa, Watson y Crick, Miller, Avery, Meselson y Stahl, Mendel, Calvin.65.- Agrupa de tres en tres, mediante una frase, los términos relacionados:
Replicación, código, anticodones, ribosomas, cadena, locus, Okazaki, ARNm, ADN, gen, semiconservativa, retardada, genético, tripletes, cromosoma, traducción, ARNt, degenerado.
33
33